
PathoGen
Pandemic calculator
Frank U. Kugelmeier - St. Ursula Grammar School, Attendorn
User manual
 | Content |
|---|---|
| 0. | Preliminary note |
| 1. | User interface |
| 1.1 | Start screen |
| 1.2 | Simulation |
| 1.3 | Data monitor |
| 1.4 | Display panels |
| 1.4.1 | Results window |
| 1.4.2 | Pathogen display |
| 1.4.3 | Run display |
| 2. | Controls |
| 2.1 | Control types |
| 2.2 | Main controls |
| 2.3 | Simulation settings |
| 2.4 | Random number navigation |
| 2.5 | Data processing keys |
| 2.6 | General program features |
| 2.6.1 | Language selection |
| 2.6.2 | External pages |
| 2.6.3 | Program settings |
| 3. | Algorithms |
| 4. | Useful lifehacks |
| 4.1 | System requirements |
| 4.2 | Known problems |
| 4.3 | Frequently asked questions |
| 5. | Bibliography |
| 6. | User licence and contact |
|
Years ago, the World Health Organisation (WHO) warned of the medical, socio-political and economic dangers of supra-regional epidemics, so-called pandemics. But it was only with the outbreak of the global COVID 19 pandemic in late autumn 2019 that this warning seems to have entered the minds of those responsible. Since then, politicians - at least in societies that are not governed by autocrats - have remarkably sought to close ranks with the relevant sciences. On the one hand, medical expertise is demanded, on the other hand, sociological diagnostics, which provide the foundation for calculating pandemic development scenarios within a population, but also worldwide. Sociological model calculations and simulations have made and continue to make a contribution to this diagnosis. In connection with the Corona pandemic, reference should be made here - only for the German-speaking countries - to the NeherLab of the University of Basel with its interactive COVID-19 simulation accessible to everyone, the COVID-19 simulator of the University of Saarland (which has been regularly used by the German government, among others), the CovidSIM model supported by the German Federal Ministry of Education and Research, and the COVID-19 simulation developed at the Institute for Information Systems Engineering of the Vienna University of Technology and used by the Austrian government. The present program PathoGen (= patho-generator) also offers such model calculations. Unlike the previously mentioned programs, however, these modellings are not limited to COVID-19, but offer an overview of the pandemic course of a wide variety of viral and bacterial pathogens. In addition to various current Corona strains, other pathogens such as measles and smallpox as well as "classics" such as Spanish flu, bubonic plague and cholera are also taken into account. The foundation of the program is an agent-based simulation, a so-called multi-agent system: many (up to one million) computer-generated "agents" with the "human" property of falling ill and being able to spread the pathogen interact in a virtual agent society and, if necessary, go through four (or five) phases of a typical course of disease: (1) Initially they are healthy but at risk of infection, then they may become infected (by chance) and go through (2) a non-infectious phase, then after a certain latency period through (3) an infectious phase (possibly but not necessarily symptomatic), which finally (4) leads either to recovery or death. The immunity to a pathogen acquired during recovery or through vaccination can, depending on the type of disease, be limited in time and in the worst case (5) lead to renewed exposure. The results of the model calculation carried out are displayed by PathoGen in three ways. The program offers
Although the program specifies (currently 24) pathogens, their properties can be changed almost at will. With the help of the implemented "adjusting screws" (sliders, switches) it is possible to generate variants of the predefined pathogens or completely new pathogens and thus also new disease processes. In this way, the program offers a multi-layered experimental field for observing and analysing pandemic developments.
|
The user interface of the program shows a large main field occupied by a diagram. Above the main field, in addition to a bar for switching various graphs on or off, there is a window that provides information about the results of the simulation process. Around these three fields - diagram field, switch bar and results window - various sliders, buttons and switches are grouped at the top, right and bottom. With their help the course of the simulation and the appearance of the program can be influenced. |
|||||||||||||||||||||
1.1 Start screen
At the beginning of the program, the main field does not initially show a diagram, but brief information. The results window is initially obscured by the PathoGen logo. It does not contain any data yet. To run a first simulation (with the default COVID-19/wild type), just press the "Start" button. If the pandemic development of another pathogen is to be simulated, the "adjusting screws" (sliders, switches) to the right of the main field can be set accordingly.
|
|||||||||||||||||||||
1.2 Simulation
As already mentioned, you can use the "Start" button to activate a (new) simulation run. A white area is now displayed on the main field, which, depending on the size of the agent population and the number of simulation days, is filled with a progression diagram either immediately or - in the case of more extensive calculations - only after a few seconds or even minutes. For longer calculations, the message "Loading..." will appear on the start button, and you should wait for it to disappear. The diagram that follows shows the number of simulation days on the x-axis and the size of the population on the y-axis. In addition, it displays different coloured history graphs that reflect various aspects of the pandemic development. The following parameters are displayed on a daily basis:
However, identical result diagrams can also be generated by switching the third navigation button to repeat mode. If the basic settings of the program remain unchanged, this is of course pointless. However, if you change the basic settings from run to run, you can use this mode to check what pandemic consequences these changes cause with defined identical coincidences. The "Compare" button can be used to switch back and forth between the current diagram and its predecessor (as well as the respective associated data sets). The "Clear" button is used to manually empty the diagram field - especially when the auto reset function is deactivated. Above the diagram field there is a results window that shows the most important results of the current simulation run in 18 parameters. The parameters correspond to the diagram curves in terms of their colouring. Further results are provided by the data monitor described in more detail below. |
1.3 Data monitor
Pressing the "Data monitor: on | off" button opens (or closes) the so-called data monitor. This records the pandemic development for each individual simulation day. The data monitor table appears at the bottom of the user interface (that is, as an extension of the program window). Its scope depends on the number of simulation days. In addition, it is possible to transfer the data to a separate window using the "Export" button. They can usually be edited better there than in the actual program window. If you call up PathoGen in the Firefox browser, you can copy and paste the data table into an external program such as MS Word (TM) and process it there. To do this, right-click on the table and select "Select all" in the drop-down box. Then, again with the right mouse button, click on the now selected table and select "Copy" in the drop-down box. You can then pass the copied table on to another program. In MS Word (TM), for example, a 100-day table exported in 9 pt fits exactly on two pages formatted with 1.5 cm top and bottom margins. The following document shows an example of a successful data transfer via Firefox.
|
| Example of a data export file | ||
|---|---|---|
1.4 Display panelsAbove the diagram field, there is a display window that documents the results of the simulation. Two additional fields are placed in the right area of the user interface, which, among other things, indicate the last selected pathogen and the current run number (i. e. random selection). |
1.4.1 Results window
The results window documents the results of the simulation run displayed in the diagram field. The parameters noted in the window are self-explanatory.
|
1.4.2 Pathogen display
|
1.4.3 Run display
The program distinguishes between "real" new runs (with new random numbers) and run repetitions (with the same random numbers). Runs with new random numbers are displayed to the left of the dot, run repetitions are displayed in a slightly smaller font to the right of the dot. For example, the display "5.3" means that the 5th run (i.e. the 5th random number selection) has been called up a total of three times. In the "blackbox" setting (see below), no runs are counted. Instead, "Run 0" is noted here. The current random number mode is displayed on the far right: |
| Each time a simulation is started, a new defined chain of random numbers (each with a new run number) is called. | |
| Each time the "Start" key is activated, the same defined random number chain is used. If you do not change any parameters compared to previous calls, the simulation run will therefore bring exactly the same result as its predecessors with the same run number. | |
| The "black box" ("run 0") means that the program does not use any defined random number chains. Instead, each simulation run uses "random random numbers" and is therefore not reproducible. |
For more details on the various random number options, see the section "Random number navigation". |
2.1 Control typesPathoGen uses four types of control elements as adjusting screws or switches for controlling the program functions. |
| Switch buttons: Depending on the function, these have the effect of (toggle) switches or push buttons. They trigger a program event. Depending on the switching status, their symbol image may vary. | |
| Sliders (range controls): They can be used to set a value (usually a numerical value) within a certain range. The current value is displayed above the slider. | |
| Single switches (checkboxes): These allow a function to be switched on (with a tick) or off (without a tick) in a simple way. | |
| Input field: A user-defined text can be entered on this field. |
2.2 Main controlsThe main control of the program is via three large keys below the diagram field. |
| Main keys | ||
|---|---|---|
| This key calls up (in text and picture) the previous simulation result. By pressing the key again, the current result is displayed again. This option is therefore suitable for comparing successive results. | ||
| This key can be used to start the calculation of the pandemic development. If the process takes a little longer - e.g. with a large population and many simulation days - the key displays the message "Loading..." during the calculation. | ||
| This key clears the current graphic display in the diagram field. However, the values of the current and previous simulation remain stored in the program, so the graph and its immediate predecessor can be restored by pressing the "Compare" key. | ||
2.3 Simulation settingsTo the right of the diagram field there are all controls and switches that are relevant for the course of a simulation. Please note: All parameters must be set before a simulation run. |
| Simulation settings | Initial value | |
|---|---|---|
| Disease type: With the PathoGen program, you can generate a variety of pandemic courses of various viral or bacterial diseases. The "Disease type" slider gives you a few "pre-programmed" pathogens to choose from. However, you can use the program's controls and switches to change a wide range of these and adapt them to your ideas. The name of the selected pre-programmed disease is noted in the display above. The following options are available:
 For the plausibility of the model calculations, please read the notes in the
"Algorithms" chapter. For the plausibility of the model calculations, please read the notes in the
"Algorithms" chapter. |
COVID-19 (wild type) |
|
| Simulation days: Here you can select for how many simulation days the pandemic development is to be calculated. The minimum is 100, the maximum 1000 days. |
200 | |
| Population: The number of agents involved can be set in steps of one hundred between 100 and 10,000. With the switch next to it, these values can be increased by a factor of 100. The population can then be selected in steps of ten thousand between 10,000 and 1,000,000.  With a very high population and a high number of simulation days, the calculation of the course
of the pandemic can take a while - on fast computers about a minute or two, on slower ones sometimes significantly longer. With a very high population and a high number of simulation days, the calculation of the course
of the pandemic can take a while - on fast computers about a minute or two, on slower ones sometimes significantly longer. |
1000 | |
| Factor 1 | 100: If the switch is set - see above - the population can be increased by a factor of 100. |
off | |
| Initial infections: It is determined here how many people are already infected at the beginning of the simulation (at the stage of exposure). The number of people can be specified in absolute numbers or percentages (percentage of the total population), depending on the status of the adjacent switch. |
10 | |
| absolute | per cent: If the switch is set, the initial infections are calculated as a percentage of the total population, otherwise in absolute numbers. |
off | |
| Spread rate: This refers to the basic reproduction number R0. The number indicates how many other people an infected person infects on average. The value can be set in tenths of a percent from 0.0 to 20.0. |
2.9 | |
| Onset of infectivity: Here you can set the day on which an exposed (non-infectious) person enters the stage of infectious disease. |
day 6 | |
| Duration of infectivity: Here you can specify how many days the infectious disease phase lasts. |
12 days | |
| Duration of immunity: The duration of immunity after recovery or vaccination (in days) can be specified here. Values from 0 to 500 days or the value "infinite" (∞) can be set. |
180 days | |
| Mortality rate: This option determines the percentage of deceased among the exposed/infected (0 to 100 per cent).  Attention. The mortality rate that can be set here is only a "guide value". The actual rate within a simulation run may vary slightly from the target
due to chance. In addition, the actual mortality rate increases when waves of infection occur as a result of dwindling immunity, since recovered or
vaccinated people are now exposed to a new infection and thus to a renewed risk of death.
Attention. The mortality rate that can be set here is only a "guide value". The actual rate within a simulation run may vary slightly from the target
due to chance. In addition, the actual mortality rate increases when waves of infection occur as a result of dwindling immunity, since recovered or
vaccinated people are now exposed to a new infection and thus to a renewed risk of death. |
6% | |
| Therapy: This controller can be used to determine the extent to which the pandemic can be countered with medical means. Two options are available: (1) If the switch to the right of the slider (see below) is set to "hospitalised" (  ),
the program creates intensive care beds in hospitals for one per mil of the population. This value can be selected in 0.1 per mil increments
from 0 to 10 per mil. It is assumed here that the patients' chances of survival can be increased by a certain factor through intensive medical
care (the factor f = 2, for example, means that the chance of survival is doubled, i.e. the risk of death is halved; the factor f = 60 sets the
chance of survival at sixty times). The level of the factor depends on the selected disease type and cannot be changed within the program.
In the case of bacterial infections, since antibiotics can be administered here, the factor is usually significantly higher than in the case
of viral diseases, for which a suitable vaccine is not always available. ),
the program creates intensive care beds in hospitals for one per mil of the population. This value can be selected in 0.1 per mil increments
from 0 to 10 per mil. It is assumed here that the patients' chances of survival can be increased by a certain factor through intensive medical
care (the factor f = 2, for example, means that the chance of survival is doubled, i.e. the risk of death is halved; the factor f = 60 sets the
chance of survival at sixty times). The level of the factor depends on the selected disease type and cannot be changed within the program.
In the case of bacterial infections, since antibiotics can be administered here, the factor is usually significantly higher than in the case
of viral diseases, for which a suitable vaccine is not always available.Incidentally, infectious patients cannot infect anyone else once they are admitted to the intensive care unit because of the safety measures in place there. The fact that the number of intensive care beds is only in the per mil range is quite realistic. For Germany, for example, the ratio of intensive care beds to population is about 0.3 per mil. Since only about one third of the intensive care beds are unoccupied, in the event of a pandemic only about 0.1 per mil beds would be available in relation to the total population. (2) If the switch is set to "outpatient" (  ), it is assumed that the pandemic
can be treated with outpatient medical means (i.e. without hospitalisation). The slider now determines how many per cent of the total population
can be reached with such a therapy. Values from 0 to 100 per cent can be set. Here, too, the chances of survival of those treated depend on a
factor f that varies depending on the pathogen. Compared to the "hospitalised" option, however, this factor is only half as high, since many
of the therapy options offered by an intensive care unit are not available at home. ), it is assumed that the pandemic
can be treated with outpatient medical means (i.e. without hospitalisation). The slider now determines how many per cent of the total population
can be reached with such a therapy. Values from 0 to 100 per cent can be set. Here, too, the chances of survival of those treated depend on a
factor f that varies depending on the pathogen. Compared to the "hospitalised" option, however, this factor is only half as high, since many
of the therapy options offered by an intensive care unit are not available at home.It should be noted that infectious patients can continue to infect other residents in their home environment, so they are not "neutralised" by outpatient care. |
0% f=2 |
|
| Therapy outpatient | hospitalised: When the switch is open (  ), the therapy option "outpatient" applies, when the
switch is closed ( ), the therapy option "outpatient" applies, when the
switch is closed ( ), the option "hospitalised" is set (see above). ), the option "hospitalised" is set (see above). |
off | |
| Vaccination rate: This slider can be used to determine what percentage of the total population will be vaccinated.  This option can only be called up if a vaccine is available for the selected pathogen. This option can only be called up if a vaccine is available for the selected pathogen. |
0% | |
| First day of vaccination: Here you can set the day on which the vaccinations are to be started. The value can be set in steps of ten from 0 (vaccination at the start of the simulation) to 1000 (vaccination on the 1000th day).  For the sake of simplicity, it is assumed that all those willing to be vaccinated can actually be
vaccinated on the first day of vaccination. It is also assumed that vaccinations are successful in 100 per cent of the cases. If you assume a
vaccination effect of less than 100 per cent, you may have to readjust the setting of the vaccination rate slider above. For the sake of simplicity, it is assumed that all those willing to be vaccinated can actually be
vaccinated on the first day of vaccination. It is also assumed that vaccinations are successful in 100 per cent of the cases. If you assume a
vaccination effect of less than 100 per cent, you may have to readjust the setting of the vaccination rate slider above. This option can also only be called up if a vaccine is available for the selected pathogen. This option can also only be called up if a vaccine is available for the selected pathogen. |
0 | |
| Multiple vaccinations: When set, the switch allows multiple vaccinations if the immunity status of those recovered or vaccinated is limited.  If no vaccine is available for the selected pathogen, the switch will be disabled.
If no vaccine is available for the selected pathogen, the switch will be disabled. |
off | |
2.4 Random number navigationThe simulated pandemic courses are based on random numbers. These are generated by PathoGen in different ways:
The advantage of the former method is that the random numbers generated in this way can be reproduced at any time. Each defined chain of random numbers is assigned to a specific run number. For example, each "run 5" will produce the same results on any computer at any time, provided the other program settings have not been changed; each "run 6" will produce new (different) but also specific and reproducible results; each "run 7" will again produce new (different) but specific results, and so on. In this way, one can repeat certain simulation runs at any time and analyze them specifically. The second method is always useful if - for example in teaching situations or in seminars - one wants to achieve random results on several computers which are not identical (i.e. which are really "differently random"). Since the simulation runs are never completely identical, comparing the results on different computers can quickly clarify, for example, the range in which the consequences of certain simulation settings move. In the run display of the program, this procedure is marked as "run 0" or "black box" mode (you cannot look into a black box). "Black box" runs are not counted. Numbering them consecutively would be meaningless, since each run is unique and thus not repeatable. The various random number selections can be controlled via four navigation buttons located to the right of the main keys. |
| Navigation buttons | Symbol | |
|---|---|---|
| The first button (left) calls the numerically following run (i.e. the following chain of random numbers). If the program is in "black box" mode, the button switches PathoGen back to the algorithm of defined random number chains and there to the "new defined random numbers" mode; in this case it has the same function as the reset button (see below). | ||
| The second button (half-left) recalls the numerically previous run (i.e. the previous chain of random numbers). If the program is in run 1, the button switches PathoGen to free random number mode ("black box" mode, "run 0"). In the "black box" mode itself, the button has no function. | ||
| The third button (half-right) switches, if the program is not in "black box" mode,
between new run (run with new defined random numbers) and run repetition (run with the same defined random numbers). The current
mode is noted in the run display. The "new" mode appears there as a dark red "New" circle, the "repeat"
mode as an arrow circle. If the program is in the "black box" mode (recognisable by a black box in the run display), the key switches to the "new" mode, i.e. to the "new defined random numbers" mode, and there back to run 1.1. This corresponds to the "reset" function (see below). |
||
| The fourth button (right) performs a reset (restart): The program is reset to run number 1.1 and to "new" mode, the diagram field is cleared, and the program logo appears in the results window. The other program settings are not reset, so that you can continue working with them immediately. | ||
If you tap the forward or backward button step by step, the run number in the run display is adjusted. However, the changed run number is only valid with the next simulation run. New runs (with new defined random numbers) are shown in the run display to the left of the dot, and repeated runs (with the same defined random numbers) are shown in slightly smaller letters to the right of the dot. |
2.5 Data processing keysThe data generated with PathoGen can be displayed in the so-called data monitor, and the program settings relevant for this can be temporarily or permanently saved and reloaded. The program provides the following keys in this context. |
| Data monitor | |
|---|---|
| The button opens ( |
|
| The "Export" button transfers the data to an external (second) window. In addition to the daily data, the results of the results window are also shown here. | |
| Backup | |
| Using this key, you can load previously saved user data (see below). The data is then available as a "custom disease type". - If no user data is available yet, an error message appears. | |
You can define any new or variants of existing pathogens using the five sliders "Spread rate", "Onset of infectivity", "Duration of infectivity",
"Duration of immunity" and "Mortality rate". With the help of the present button the values of these controllers as well as the intensive care factor f, the
pathogen type (virus or bacterium) and a disease name can be stored, namely
 Each press of the key deletes the previously saved user-defined values of the program without warning and replaces them with the current ones.
Each press of the key deletes the previously saved user-defined values of the program without warning and replaces them with the current ones. The data storage options depend on the browser type and its settings. If necessary, test out several browsers.
The data storage options depend on the browser type and its settings. If necessary, test out several browsers. |
|
You can use this button - after a security query - to delete permanently saved user-defined values without replacing them with
new ones. The user settings temporarily contained in the program are not affected by this. - If there is no saved user data, an error message appears. When the program starts, the key is hidden for security reasons. It only appears on the right below the monitor list when the data monitor is open.
When the program starts, the key is hidden for security reasons. It only appears on the right below the monitor list when the data monitor is open. |
|
2.6 General program featuresAt the top and bottom of the user interface, but also above the diagram field, there are various buttons, switches and controls that do not determine the simulation itself, but the layout of the program or activate external pages. |
2.6.1 Language selection |
| Language selection | ||
|---|---|---|
PathoGen can be accessed in the following languages:
However, this manual is currently only available in German and in English. |
||
2.6.2 External pages |
| External pages | ||
|---|---|---|
| This button leads back to the index page. | ||
| This button can be used to open the present manual in a separate window. | ||
2.6.3 Program settings |
| Diagram switches | Initial value | |
|---|---|---|
| Using the diagram switches arranged above the diagram field, individual curves of the diagram as well as the vaccination marker can be switched on or off. | ||
| Susceptible: This switch can be used to turn on or off the green "Susceptible" (= healthy but at risk of infection) curve. |
on | |
| All cases: This switch can be used to switch on or off the black "All cases" (= sum of all those exposed or infected so far) curve. |
on | |
| Active cases: This switch can be used to switch on or off the red "Active cases" (= currently exposed or infected) curve. |
on | |
| Immunised: This switch can be used to switch on or off the purple "Immunised" (= recovered and vaccinated within the immunity period) curve. |
on | |
| Deceased: This switch can be used to switch on or off the blue "Deceased" (= died of the pathogen) curve. |
on | |
Utilisation:
 Via this switch, the ochre-coloured curve showing the degree of utilisation of the hospital intensive care units can be switched on or off. The curve shows the degree of utilisation in per cent (0 to 100). However, it is only displayed if the "Therapy" switch has been set to "hospitalised" for the simulation. Please click on the image to enlarge it. |
on | |
| Vaccination: With this switch the turquoise vaccination marker(s) can be switched on or off. |
on | |
| Further diagram field settings | Initial value | |
| Line width: Here you can set the width of the lines in the diagram field in a grid from 1 to 10. - Attention: The width has no effect on the simulation process. It is only used for better visualisation on small computer displays. |
4 | |
| Graphic grid off | on: Use this option to show ( |
on | |
| Auto reset: If this switch is open, each graph is retained on the diagram field, even when the next simulation run is carried out. In this way, you can superimpose several pandemic courses and, for example, determine the scatter of certain settings. - When the switch is closed, the previous graph is automatically deleted at the beginning of each simulation run. |
on | |
| Vaccination marker: With this switch the appearance and position of the vaccination mark(s) on the diagram field can be determined. With the switch open, vaccination marks are placed as a turquoise vertical dash ( | ) starting from the bottom of the diagram. If the switch is closed, a corresponding triangle (▼) appears at the top of the diagram instead. The following are examples of both variants. 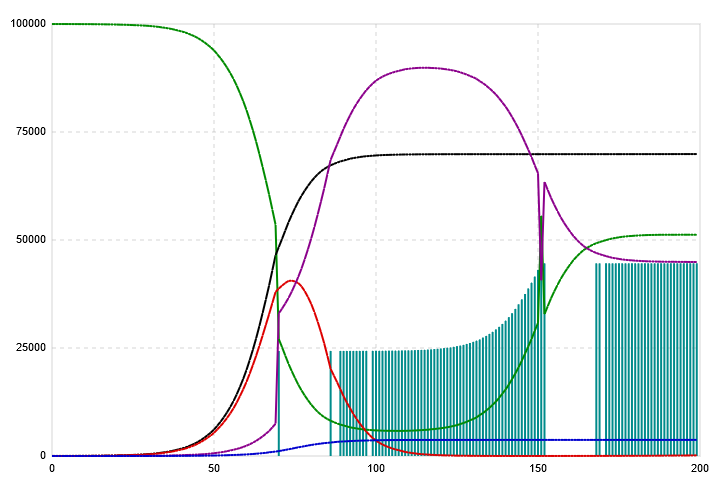 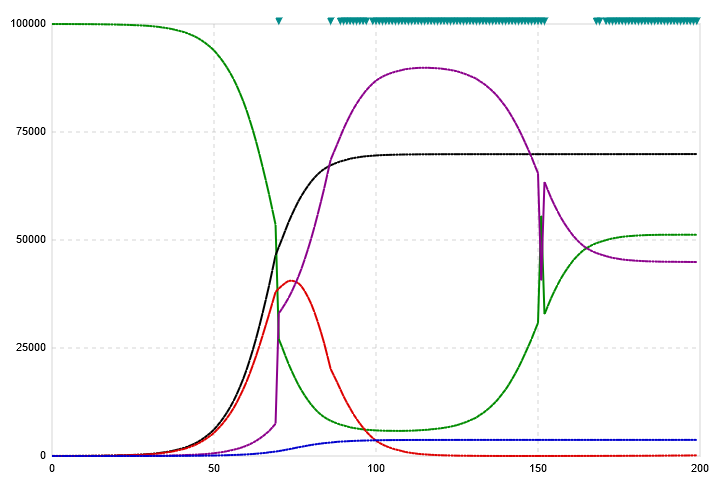 Please click on the images to enlarge them. |
on | |
| Backup settings | Initial value | |
| Data backup: If this switch is open, user-defined settings can only be saved temporarily (i.e. until the end of the program). If the switch is closed, the settings can also be saved permanently on your computer. However, you must give your express consent for this. When you close the switch for the first time, a confirmation prompt appears. |
off | |
| Disease type input field: This field allows you to give the user-defined disease a name of up to twenty characters. The input is confirmed by a mouse click in the field or by using the Enter key. After confirmation, the program jumps to the disease type "custom".  The input field is designed for twenty average proportional characters. If you use a lot of unusually wide characters, it can happen that
the name "breaks up" the program's display windows. In this case, please reduce the number of characters.
The input field is designed for twenty average proportional characters. If you use a lot of unusually wide characters, it can happen that
the name "breaks up" the program's display windows. In this case, please reduce the number of characters.
|
empty | |
| Data monitor settings | Initial value | |
| Data collection off | on: If this switch is open, the program does not generate any data for the data monitor, which means that no data can be subsequently retrieved for detailed analysis. It only makes sense to switch off data collection in order to increase the computing capacity - for example on older computers - and to speed up calculations. - When the switch is closed, data collection is activated. |
on | |
| Font size for data export: The table generated by the data monitor can be called up in different font sizes in the export version. The size can be set here from 8pt to 12pt. |
11pt | |
| Latest data top | bottom: This option allows you to toggle the direction of data display. When the switch is open ( |
on | |
| Design setting | Initial value | |
| By (repeatedly) pressing this button, the colour design of the user interface can be varied. Ten colour variants are available. Please note that the actual design shown depends on the browser. | 1/10 | |
|
The disease progression on which the program is based follows the SEIR or SEIRS model for describing the spread of infectious diseases (SEIR: susceptible, exposed, infectious, recovered/removed; with SEIRS a new susceptible phase is added). It is therefore assumed that a susceptible (= person at risk) first becomes pre-infected without being infectious (i.e. he is only "exposed"). After this latency period, as soon as he is infectious, he is considered to be "infected" (= infectiously ill, although symptoms may or may not accompany this illness). After that, two possibilities open up: Either he dies - or he recovers after the infectiousness has subsided and is immunised (at least for a while) (= SEIR). If his immunity wanes, however, he falls back into the stage of being at risk (= SEIRS). Depending on the pathogen, the key data of the disease (spread, latency period, infectivity, mortality, immunity) vary greatly. The same applies to the success of intensive medical care. As a rule, bacterial diseases can be better treated medically (e.g. with antibiotics) than viral infections, as there are often no adequate vaccines against the latter. The PathoGen program currently takes into account 24 different pathogens whose key data are predefined by the software. These data correspond to the current findings of relevant scientific publications, which are listed in the bibliography. With a view to future virus variants or completely new virus or bacterial strains, however, the values can be largely changed or adapted via the corresponding sliders. The following table gives an overview of the implemented diseases and their set parameters. The following are taken into account:
|
| a | b | c | d | e | f | g | h | i |
|---|---|---|---|---|---|---|---|---|
| COVID-19 (wild type) | 2.9 | 6 | 12 | 180 | 6.0 | 4 | virus | yes |
| COVID-19 (Delta) | 6.9 | 4 | 12 | 180 | 8.0 | 4 | virus | yes |
| COVID-19 (Omicron) | 9.5 | 6 | 12 | 180 | 1.5 | 4 | virus | yes |
| SARS-CoV-1 2002 | 3.5 | 6 | 12 | 180 | 11.0 | 2 | virus | no |
| MERS-CoV 2012 | 0.8 | 10 | 10 | 200 | 34.0 | 2 | virus | no |
| Spanish flu | 2.5 | 2 | 4 | 180 | 10.0 | 2 | virus | no |
| Swine flu | 1.5 | 2 | 4 | 180 | 0.5 | 2 | virus | yes |
| Seasonal flu | 1.5 | 2 | 11 | 350 | 0.5 | 5 | virus | yes |
| Flu-like infection | 2.5 | 1 | 10 | 45 | 0.0 | 1 | virus | no |
| Ebola fever | 2.0 | 8 | 19 | ∞ | 59.0 | 10 | virus | yes |
| Marburg virus | 1.3 | 6 | 16 | ∞ | 50.0 | 2 | virus | no |
| Lassa fever | 1.5 | 8 | 15 | 30 | 5.0 | 5 | virus | no |
| Measles | 15.0 | 10 | 6 | ∞ | 1.0 | 4 | virus | yes |
| Rubella (postnatal) | 6.0 | 17 | 14 | 500 | 0.0 | 1 | virus | yes |
| Mumps | 5.5 | 17 | 14 | ∞ | 0.0 | 1 | virus | yes |
| Chickenpox | 11.0 | 15 | 10 | ∞ | 0.0 | 1.1 | virus | yes |
| Smallpox (Variola) | 6.0 | 13 | 15 | ∞ | 30.0 | 20 | virus | yes |
| Monkeypox | 0.6 | 13 | 21 | ∞ | 1.0 | 4 | virus | yes |
| Scarlet fever | 1.5 | 2 | 19 | 500 | 0.5 | 30 | bact. | no |
| Whooping cough | 6.0 | 12 | 35 | ∞ | 0.5 | 30 | bact. | yes |
| Diphteria 1880 | 3.0 | 4 | 21 | 500 | 50.0 | 3.3 | bact. | yes |
| Bubonic plague | 3.0 | 4 | 4 | 180 | 60.0 | 20 | bact. | yes |
| Pneumonic plague | 3.0 | 2 | 5 | 20 | 95.0 | 4 | bact. | yes |
| Cholera | 9.5 | 2 | 5 | 130 | 60.0 | 60 | bact. | yes |
| custom | 2.9 | 6 | 12 | 180 | 6.0 | 4 | virus | yes |
|
The "custom" setting (as the 25th option) corresponds to the values of COVID-19 (wild type) when the program starts, but can be changed and saved as desired.
(1) The program does not provide exact medical forecasts, but merely maps pandemic development trends. As a rule, it generates the maximum possible number of cases, since it is assumed as a model that infectious agents can actually infect their fellow residents unhindered during the entire period of their contagiousness. In practice, however, this is not the case. Symptomatically ill people withdraw from the public sphere in a debilitated state or are quarantined, so that the number of their outside contacts and thus the transmission of the pathogen is drastically reduced. The pandemic calculator thus basically reproduces above all those - tendentially precarious - forms of society in which such ("voluntary" or forced) withdrawal is not possible. (2) In addition, the program only considers outpatient or intensive care as a factor in attenuating infections. Other factors for pandemic control, such as special hygiene rules, travel or contact bans, the closure of public facilities or even a complete lockdown, are left out here. If you are particularly interested in these latter measures, please consult the related program Pandemic Laboratory. (3) PathoGen also focuses on the model idea with regard to the implemented vaccination option. Two ideal-typical conditions are assumed: all desired vaccinations (a) can be carried out on a single simulation day and (b) basically achieve 100% vaccination protection. Here, too, the practice unfortunately looks different. Nevertheless, the effectiveness of the vaccinations can be indirectly put into perspective via the "vaccination rate" slider. (4) The SEIR model on which the program is based assumes complete recovery (or death) after an illness. Permanent damage or secondary diseases, i.e. phenomena such as Long Covid, are not represented by this. (5) Not all infectious diseases are transmitted from person to person by direct contact (droplet or smear infection). Many diseases, such as malaria, dengue fever or Lyme disease, can be traced back to transmission through animals, such as mosquitoes and ticks. The latter diseases are not included in the present program, as completely different algorithms (which, for example, take into account the climatic and seasonal conditions that are indispensable for the animals' habitat) would have to be used. (6) The only exception is bubonic plague (included in the program), since its carriers (rat fleas) live in close symbiosis with humans, regardless of climate and region, and direct infection is thus possible, for example via human clothing (i.e. quasi-body contact). (7) The simulation of cholera (also included here) proves to be problematic. Modelling this disease using the SEIR(S) model is only possible to a limited extent, since transmission rarely occurs directly from person to person, but usually indirectly via contaminated water, faeces or contaminated food. SEIR(S)-compatible modelling, as undertaken here by the program, actually only applies to populations in decidedly precarious living conditions (living together in very confined spaces under poor hygienic conditions). (8) The important diseases tuberculosis and HIV/AIDS are not included, as their course of infection defies simple modelling in the SEIR(S) model. In the case of tuberculosis, the latency period can be a few weeks or months, but can also extend over several years; in most exposed persons (about 9 out of 10), the disease even never breaks out. The situation is similar for HIV; in addition, patients usually do not die from HIV itself, but from one of numerous (diffuse, not modelled here) secondary diseases (e.g. mycobacterial infections [B20.0] such as tuberculosis, or pneumocystis pneumonia [B20.6]). (9) Reliable, unambiguous data are not available for all of the diseases listed in the above table. These vary considerably, in part, depending on the period under study and the region. For historical pandemics (bubonic plague, diphtheria of the 1880s, Spanish flu, etc.) only estimated values are available anyway. For example, estimates of the number of deaths caused by the Spanish flu (1918-1920) vary between 20 and 50 million worldwide, depending on the scientific publication. Some of the figures used in the program are therefore only approximate. (10) In part, the key data determined for a disease also vary greatly because, of course, not all patients react in the same way to a pathogen. Therefore, (weighted) average values are often set in the table and in the program. For example, the latency period for COVID-19 (wild type) lies in a range of two to fourteen days, but typically the infectious disease sets in after just under a week. The onset of infectiousness is therefore indicated in the table with the "fixed" value 6 (= 6th day) and is also calculated in the same way in the program. (11) For the sake of simplicity, the model used in PathoGen equates the immunity of recovered and vaccinated persons. In practice, however, there can be significant deviations here. For example, after an actual infection with measles or mumps, patients are usually immune for life; however, (approximately) comparable protection is only achieved with a double vaccination. (12) For some diseases, especially novel ones such as COVID-19, the duration of subsequent immunity cannot (yet) be precisely determined. Occasionally, mutated variants of the pathogen will cancel out an existing immunity. Taking these variants into account, the program currently assumes an immunity of about half a year (180 days) for SARS-CoV-2, for example. Diffuse influenza (with over 200 viral variants) also requires annual re-targeted vaccination. (13) The effectiveness of intensive care is difficult to determine in some cases, as only mitigated, but not "natural" death rates are available for medically well-supplied regions. In order to measure the effectiveness of intensive care, one would actually have to compare a group of patients receiving care with a control group of uncared for patients. Of course, this is not justifiable for ethical reasons. (14) Sometimes medical and legal claims also conflict. In the early 20th century, for example, vaccinations were given against bubonic plague; even today, plague vaccinations (whose effect is disputed) are offered. The German Robert Koch Institute, on the other hand, currently reports that there is no approved plague vaccine. (15) The PathoGen software does not claim to be absolute. If you have other key medical data than those specified in the program, you should make use of the option of adjusting the values using the adjusting screws (controllers, switches) as you wish. |
In the following section you will find some technical information that may be helpful for the optimal use of the program. 4.1 System requirementsPathoGen is programmed in JavaScript and can therefore be run on many platforms. The program was successfully tested on MS Windows (TM), macOS (TM) and Android (TM) on the following browsers:
Known limitations:
4.2 Known problems(1) On older computers, you should not immediately set the population to the highest level (1 million), as otherwise the hardware may only be able to cope with the complex calculations of the program with considerable time expenditure or not at all. So start with a moderate number of people (program default: 1000) and test the possibilities of your computer step by step. (2) A problem-free "real" transfer of the data via data export that goes beyond pure display is currently only possible with Firefox. In the other browsers, the formatted export table is opened properly in an external window, but it cannot be transferred from there, or only in a roundabout way, in its original formatting via copy & paste into external programs such as MS Word (TM). (3) If the simulation does not run satisfactorily on an older computer or in certain network environments, you can switch off the data collection option if you do not need it. This deactivates the routine for providing the monitor data internally in the program, which in turn saves computing capacity and thus perhaps improves the run. (4) The possibility of permanently storing simulation data on one's own computer depends on the type of browser used and its settings. Data is not stored via cookies, but via the Javascript function localStorage. Over time, some browsers have already changed the handling of this privacy-sensitive feature; further changes cannot be ruled out. 4.3 Frequently asked questionsEven if I keep the settings of the program, the individual simulation runs show different results. How can this be explained? The results show that reality is not completely predictable. The program only provides probabilities that the results are within a certain range. I am puzzled by the fact that the simulation results are repeatable. So are these not "accidental" at all, but rather "programmed", possibly manipulated results? All simulation results are random. The mode in which defined random number chains are used should not be imagined as having ready-made numbers. Rather, they are generated in a very complex way and diverge even with minimally changed simulation settings. The term "random number chains" therefore describes a multi-dimensional network of random numbers. Nothing is manipulated here. The random numbers are calculated using an algorithm developed by Makoto Matsumoto, Takuji Nishimura and Sean McCullough. The source code for the PathoGen program is open; the programming can therefore be checked in detail. - By the way, the results generated in the "random random numbers" mode do not differ from those in the defined random numbers mode. Isn't the model behind the program too simple? In reality, the spread of infections depends on many different factors, such as the age of the people involved, their reference groups, the population density in general, but above all the medical infrastructure. That is correct. PathoGen is based on a very simple model with very reduced infection pathways. A more complex simulation that takes your objections into account can be found, for example, in the pandemic program Pandemic Laboratory. The custom disease type defaults to the "virus" trait. How can I change this to "bacterium"? Very easily. Select a bacterial pathogen, for example diphtheria, bubonic plague or cholera, adjust the settings there to suit your needs and then save the whole thing (via the "Save" button). You can then invoke your newly created pathogen as a bacterium using the "custom" option. How can the intensive care factor be adapted to my ideas? Limited only. Choose a preset pathogen whose factor most closely matches your ideas, and then adjust the settings of that disease type to suit your needs. Unfortunately, you cannot change the factor itself. A factor f = 60 can therefore be generated via the pathogen cholera, but a factor f = 23 or f = 9 cannot. If I switch on intensive medical therapy, it can happen that no one falls ill during the entire simulation run. How is this possible? If the number of people exposed at the start of the program is lower than the number of intensive care beds provided, all infected people are of course admitted to hospital immediately when infectiousness breaks out. There they can no longer infect anyone else. The pandemic is thus over before it has even begun. Every time I want to name a custom disease based on a pre-programmed pathogen, the program jumps to the disease type "custom" and my controller settings are lost. What to do? First save your individual controller settings, then enter the name of the disease and finally save the whole thing again. Now everything should be saved. My user data cannot be saved permanently, although I have set the "Backup" switch. What could be the reason? There can be several reasons for this. The data is not saved via cookies, but via the Javascript function localStorage. However, perhaps your browser is set to prohibit both the use of cookies and localStorage. In this case, you will need to change your browser settings (e.g., allow cookies). Your browser may also block the handling of localStorage in certain working environments, for example, if you have downloaded the PathoGen program locally to your computer and use it offline. In case of difficulties, please try different browser types. What additional features are planned for the program in the future? PathoGen is deliberately kept simple and only models pandemic events in very general terms. It should stay that way. If you are interested in more complex pandemic simulations, please use the Pandemic Laboratory. This offers very sophisticated setting options and even more extensive options for data analysis. |
A notification of the original author in case of non-trivial modification of the program is explicitly desired. Changes to the texts of the manual please only after consultation with the author. If you have any questions, please contact the following address:
|
| English text created by automatic translator. Version 3 |
 | Inhalt |
|---|---|
| 0. | Vorbemerkung |
| 1. | Die Benutzeroberfläche |
| 1.1 | Der Startbildschirm |
| 1.2 | Die Simulation |
| 1.3 | Der Datenmonitor |
| 1.4 | Die Anzeigefelder |
| 1.4.1 | Das Ergebnisfenster |
| 1.4.2 | Die Pathogenanzeige |
| 1.4.3 | Die Laufanzeige |
| 2. | Die Steuerelemente |
| 2.1 | Bedienelemente |
| 2.2 | Die Hauptsteuerung |
| 2.3 | Simulationseinstellungen |
| 2.4 | Die Zufallszahlen-Navigation |
| 2.5 | Die Datenverarbeitungs-Tasten |
| 2.6 | Allgemeine Programmfunktionen |
| 2.6.1 | Die Sprachauswahl |
| 2.6.2 | Externe Seiten |
| 2.6.3 | Programmeinstellungen |
| 3. | Die Algorithmen |
| 4. | Tipps & Tricks |
| 4.1 | Systemvoraussetzungen |
| 4.2 | Bekannte Probleme |
| 4.3 | Häufig gestellte Fragen |
| 5. | Literaturverzeichnis |
| 6. | Nutzungsrechte und Kontakt |
|
Schon vor Jahren warnte die Weltgesundheitsorganisation WHO vor den medizinischen, sozialpolitischen und ökonomischen Gefahren überregional auftretender Epidemien, sogenannter Pandemien. Doch erst mit dem Ausbruch der globalen COVID-19-Pandemie im Spätherbst 2019 scheint diese Warnung in den Köpfen der Verantwortlichen angekommen zu sein. Seither sucht die Politik - zumindest in nicht autokratisch regierten Gesellschaften - in bemerkenswerter Weise den Schulterschluss mit den einschlägigen Wissenschaften. Eingefordert werden zum einen medizinische Expertise, zum anderen soziologische Diagnostik, die den Grundstein dafür liefern, pandemische Entwicklungsszenarien innerhalb einer Bevölkerung, aber auch weltweit vorauszuberechnen. Einen Beitrag zu dieser Diagnostik leisteten und leisten soziologische Modellrechnungen und Simulationen. Hingewiesen sei hier im Zusammenhang mit der Corona-Pandemie - nur für den deutschsprachigen Raum - auf das NeherLab der Universität Basel mit seiner jedermann zugänglichen interaktiven COVID-19-Simulation, den COVID-19-Simulator der Universität des Saarlandes (auf den u. a. die deutsche Bundesregierung regelmäßig zurückgegriffen hat), das vom deutschen Bundesministerium für Bildung und Forschung unterstützte Modell CovidSIM sowie die am Institut für Information Systems Engineering der TU Wien entwickelte und von der österreichischen Regierung genutzte COVID-19-Simulation. Auch das vorliegende Programm PathoGen (= Patho-Generator) bietet derartige Modellrechnungen. Anders als die zuvor genannten Programme beschränken sich diese Modellierungen jedoch nicht auf COVID-19, sondern bieten einen Überblick über den pandemischen Verlauf unterschiedlichster viraler und bakterieller Pathogene. Neben diversen aktuellen Corona-Stämmen werden auch andere Erreger wie Masern und Pocken sowie "Klassiker" wie die Spanische Grippe, die Beulenpest und die Cholera berücksichtigt. Das Programm basiert auf einer agentengestützten Simulation, einem sogenannten Multi-Agenten-System: Viele (bis zu eine Million) computergenerierte "Agenten" mit der "menschlichen" Eigenschaft, zu erkranken und den Krankheitserreger weiterverbreiten zu können, wirken in einer virtuellen Agentengesellschaft zusammen und durchleben gegebenenfalls vier (oder fünf) Phasen eines typischen Krankheitsverlaufs: (1) Anfangs sind sie gesund, doch ansteckungsgefährdet, dann infizieren sie sich möglicherweise (zufallsbedingt) und durchleben (2) eine nicht infektiöse Phase, dann nach einer gewissen Latenzzeit (3) eine (eventuell, aber nicht zwingend symptomatische) infektiöse Phase, die abschließend (4) entweder zur Genesung oder zum Tod führt. Die in der Genesung oder durch eine Impfung erworbene Immunität gegenüber einem Pathogen kann, je nach Krankheitstyp, zeitlich begrenzt sein und schlimmstenfalls (5) zu einer neuerlichen Gefährdung führen. Die Ergebnisse der durchgeführten Modellrechnung werden von PathoGen auf dreifache Weise angezeigt. Das Programm bietet
Das Programm gibt zwar (zurzeit 24) Pathogene vor, doch können deren Eigenschaften nahezu beliebig geändert werden. Mithilfe der implementierten "Stellschrauben" (Schieberegler, Schalter) ist es möglich, Varianten der vorgegebenen oder aber völlig neue Krankheitserreger und damit auch neuartige Krankheitsverläufe zu generieren. Auf diese Weise bietet das Programm ein vielschichtiges Experimentalfeld zur Beobachtung und Analyse pandemischer Entwicklungen.
|
Die Benutzeroberfläche des Programms weist ein großes, mit einem Diagramm belegtes Hauptfeld auf. Über dem Hauptfeld befindet sich außer einer Leiste zur Ab- oder Zuschaltung diverser Graphen ein Ergebnisfenster, das Auskunft über die Resultate des Simulationsverlaufs gibt. Um diese drei Felder - Diagrammfeld, Schaltleiste und Ergebnisfenster - herum gruppieren sich oben, rechts und unten diverse Regler, Tasten und Schalter, mit deren Hilfe der Simulationsverlauf und das Aussehen des Programms beeinflusst werden können. |
|||||||||||||||||||||
1.1 Der Startbildschirm
Zu Beginn des Programms wird im Hauptfeld zunächst kein Diagramm, sondern eine Kurzinformation angezeigt. Das Ergebnisfenster wird anfangs durch das PathoGen-Logo verdeckt. Es enthält noch keine Daten. Um eine erste Simulation (mit der Vorgabe COVID-19/Wildtyp) durchzuführen, genügt ein Druck auf die Taste "Starten". Soll die pandemische Entwicklung eines anderen Pathogens simuliert werden, können die "Stellschrauben" (Schieberegler, Schalter) rechts neben dem Hauptfeld entsprechend angepasst werden.
|
|||||||||||||||||||||
1.2 Die Simulation
Mithilfe der Taste "Starten" lässt sich, wie bereits erwähnt, ein (neuer) Simulationslauf aktivieren. Auf dem Hauptfeld wird nun eine weiße Fläche angezeigt, die sich, je nach Größe der Agentenpopulation und nach Anzahl der Simulationstage, entweder sofort oder - bei umfangreicheren Berechnungen - auch erst nach ein paar Sekunden oder gar Minuten mit einem Verlaufsdiagramm füllt. Bei längeren Berechnungen erscheint auf der Starttaste die Mitteilung "In Arbeit...", deren Verschwinden man getrost abwarten sollte. Das Diagramm, das sich anschließend zeigt, weist auf der x-Achse die Zahl der Simulationstage, auf der y-Achse die Größe der Population aus. Darüber hinaus zeigt es verschiedenfarbige Verlaufsgraphen an, die diverse Aspekte der pandemischen Entwicklung widerspiegeln. Folgende Parameter werden tagesaktuell dargestellt:
Allerdings lassen sich auch identische Ergebnisdiagramme erzeugen, indem man den dritten Navigationsknopf in den Wiederholungs-Modus schaltet. Bei unveränderten Grundeinstellungen des Programms ist dies natürlich sinnlos. Verändert man die Grundeinstellungen jedoch von Lauf zu Lauf, so kann man in diesem Modus überprüfen, welche pandemischen Folgen diese Veränderungen bei definiert gleichen Zufällen hervorrufen. Mit der Taste "Vergleichen" kann zwischen dem aktuellen Diagramm und seinem Vorgänger (sowie den jeweils zugehörigen Datensätzen) hin- und hergeschaltet werden. Die Taste "Löschen" dient dazu, ein Diagrammfeld - besonders bei deaktivierter Auto-Reset-Funktion - manuell zu leeren. Über dem Diagrammfeld befindet sich ein Ergebnisfenster, das in 18 Parametern die wichtigsten Resultate des aktuellen Simulationslaufs ausweist. Die Parameter entsprechen in ihrer Farbgebung inhaltlich den Diagrammkurven. Weitere Ergebnisse liefert der im Folgenden genauer beschriebene Datenmonitor. |
1.3 Der Datenmonitor
Ein Druck auf die Taste "Datenmonitor: ein | aus" öffnet (bzw. schließt) den sogenannten Datenmonitor. Dieser verzeichnet die pandemische Entwicklung für jeden einzelnen Simulationstag. Die Tabelle des Datenmonitors erscheint am unteren Rand der Benutzeroberfläche (das heißt: in Verlängerung des Programmfensters). Ihr Umfang hängt von der Anzahl der Simulationstage ab. Zusätzlich ist es möglich, die Daten über die "Export"-Taste in ein separates Fenster zu übertragen. Dort können sie in der Regel besser nachbearbeitet werden als im eigentlichen Programmfenster. Wenn Sie PathoGen im Firefox-Browser aufrufen, können Sie die Datentabelle per Copy & Paste in ein externes Programm, etwa MS Word (TM), übertragen und dort weiterverarbeiten. Klicken Sie dazu mit der rechten Maustaste auf die Tabelle und wählen Sie in der aufklappenden Box "Alles auswählen". Klicken Sie dann, wieder mit der rechten Maustaste, auf die nunmehr markierte Tabelle und wählen Sie in der aufklappenden Box "Kopieren". Anschließend können Sie die kopierte Tabelle in ein anderes Programm weiterreichen. In MS Word (TM) passt eine in 9 pt exportierte 100-Tage-Tabelle beispielsweise genau auf zwei mit 1,5 cm oberem und unterem Rand formatierte Seiten. Ein Beispiel für einen gelungenen Datentransfer per Firefox zeigt das folgende Dokument.
|
| Beispiel einer Datenexport-Datei | ||
|---|---|---|
1.4 Die AnzeigefelderÜber dem Diagrammfeld befindet sich ein Anzeigefenster, das die Ergebnisse der Simulation dokumentiert. Im rechten Bereich der Benutzeroberfläche sind zwei weitere Felder platziert, die unter anderem das zuletzt ausgewählte Pathogen und die aktuelle Laufnummer (Zufallsauswahl) ausweisen. |
1.4.1 Das Ergebnisfenster
Das Ergebnisfenster dokumentiert die Resultate des im Diagrammfeld angezeigten Simulationslaufs. Die im Fenster notierten Parameter sind selbsterklärend.
|
1.4.2 Die Pathogenanzeige
|
1.4.3 Die Laufanzeige
Das Programm unterscheidet zwischen "echten" neuen Läufen (mit neuen Zufallszahlen) und Laufwiederholungen (mit denselben Zufallszahlen). Läufe mit neuen Zufallszahlen werden links vom Punkt, Laufwiederholungen in etwas kleinerer Schrift rechts vom Punkt dargestellt. Die Anzeige "5.3" bedeutet also zum Beispiel, dass der 5. Lauf (d. h. die 5. Zufallszahlenauswahl) insgesamt dreimal aufgerufen worden ist. In der Einstellung "Blackbox" (siehe unten) werden keine Läufe gezählt. Notiert wird hier stattdessen "Lauf 0". Rechts außen wird der aktuelle Zufallszahlenmodus angezeigt: |
| Bei jedem Start einer Simulation wird eine neue definierte Kette von Zufallszahlen (mit jeweils neuer Laufnummer) aufgerufen. | |
| Bei jeder Aktivierung der Taste "Starten" wird dieselbe definierte Zufallszahlenkette verwendet. Ändert man gegenüber früheren Aufrufen keine Parameter, bringt der Simulationslauf also exakt dasselbe Ergebnis wie seine Vorgänger mit derselben Laufnummer. | |
| Die "Blackbox" ("Lauf 0") bedeutet, dass das Programm keine definierten Zufallszahlenketten verwendet. Jeder Simulationslauf benutzt stattdessen "zufällige", stets neue Zufallszahlen und ist damit nicht reproduzierbar. |
Näheres zu den verschiedenen Zufallszahlen-Optionen bietet der Abschnitt "Zufallszahlen-Navigation". |
2.1 BedienelementeAls Stellschrauben bzw. Schalter zur Steuerung der Programmfunktionen verwendet PathoGen vier Typen von Bedienelementen. |
| Schaltknöpfe: Diese haben, je nach Funktion, die Wirkung von (Wechsel-)Schaltern oder Tastern. Sie lösen ein Programmereignis aus. Je nach Schaltzustand kann ihr Symbolbild variieren. | |
| Schieberegler (Bereichsregler): Mit ihnen lässt sich ein Wert (meist ein Zahlenwert) innerhalb eines bestimmten Bereichs einstellen. Der aktuelle Wert wird über dem Regler angezeigt. | |
| Einfach-Schalter (Checkboxes): Hiermit lässt sich auf einfache Weise eine Funktion zu- (mit Häkchen) oder abschalten (ohne Häkchen). | |
| Eingabefeld: Auf diesem Feld kann ein benutzerdefinierter Text eingetragen werden. |
2.2 Die HauptsteuerungDie hauptsächliche Steuerung des Programms erfolgt über drei große Tasten unterhalb des Diagrammfelds. |
| Haupttasten | ||
|---|---|---|
| Diese Taste ruft (in Text und Bild) das vorherige Simulationsergebnis auf. Durch neuerlichen Tastendruck wird wieder das aktuelle Ergebnis angezeigt. Die Option eignet sich also zum Vergleich aufeinander folgender Resultate. | ||
| Über diese Taste lässt sich die Berechnung der pandemischen Entwicklung starten. Dauert der Vorgang - etwa bei großer Bevölkerungszahl und vielen Simulationstagen - etwas länger, wird während der Berechnung auf der Taste der Hinweis "In Arbeit..." angezeigt. | ||
| Mit dieser Taste wird die aktuelle grafische Anzeige im Diagrammfeld gelöscht. Die Werte der aktuellen und der vorherigen Simulation bleiben jedoch im Programm gespeichert, so dass die Grafik und ihre unmittelbare Vorgängerin über das Drücken der Taste "Vergleichen" wiederhergestellt werden können. | ||
2.3 SimulationseinstellungenRechts neben dem Diagrammfeld befinden sich alle Regler und Schalter, die für den Verlauf einer Simulation relevant sind. Beachten Sie bitte: Sämtliche Parameter müssen vor einem Simulationslauf festgelegt werden. |
| Simulationseinstellungen | Startwert | |
|---|---|---|
| Krankheitstyp: Mithilfe des Programms PathoGen können Sie vielfältige pandemische Verläufe verschiedener viral oder bakteriell verursachter Krankheiten generieren. Der Regler "Krankheitstyp" stellt Ihnen einige "vorprogrammierte" Pathogene zur Auswahl; diese können Sie jedoch über die Regler und Schalter des Programms in weiten Bereichen abändern und Ihren Vorstellungen anpassen. Der Name der ausgewählten vorprogrammierten Krankheit wird im darüber befindlichen Anzeigefeld notiert. Zur Wahl stehen folgende Optionen:
 Lesen Sie zur Plausibilität der Modellrechnungen bitte die Anmerkungen im Kapitel
"Die Algorithmen". Lesen Sie zur Plausibilität der Modellrechnungen bitte die Anmerkungen im Kapitel
"Die Algorithmen". |
COVID-19 (Wildtyp) |
|
| Simulationstage: Hier kann gewählt werden, für wie viele Simulationstage die pandemische Entwicklung berechnet werden soll. Das Minimum sind 100, das Maximum 1000 Tage. |
200 | |
| Population: Die Zahl der beteiligten Agenten lässt sich in Hunderterschritten zwischen 100 und 10.000 einstellen. Mit dem danebenliegenden Schalter können diese Werte um den Faktor 100 erhöht werden. Die Population ist dann in Zehntausenderschritten zwischen 10.000 und 1.000.000 zu wählen.  Bei sehr hoher Bevölkerungszahl und einer hohen Zahl an Simulationstagen kann die
Berechnung des Pandemieverlaufs eine Weile dauern - auf schnellen Rechnern etwa ein, zwei Minuten, auf langsameren teils deutlich länger. Bei sehr hoher Bevölkerungszahl und einer hohen Zahl an Simulationstagen kann die
Berechnung des Pandemieverlaufs eine Weile dauern - auf schnellen Rechnern etwa ein, zwei Minuten, auf langsameren teils deutlich länger. |
1000 | |
| Faktor 1 | 100: Bei gesetztem Schalter kann - siehe oben - die Population um den Faktor 100 erhöht werden. |
aus | |
| Infektionen zu Beginn: Festgelegt wird hier, wie viele Personen zu Beginn der Simulation bereits (im Stadium der Exposition) infiziert sind. Die Personenzahl kann, je nach Schaltzustand des nebenstehenden Schalters, in absoluten Zahlen oder Prozentwerten (Anteil an der Gesamtpopulation in Prozent) angegeben werden. |
10 | |
| absolut | prozentual: Bei gesetztem Schalter werden die anfänglichen Infektionen prozentual zur Gesamtpopulation, ansonsten in absoluten Zahlen berechnet. |
aus | |
| Ausbreitungsrate: Gemeint ist die Basisreproduktionszahl R0. Die Zahl gibt an, wie viele andere Menschen eine infizierte Person durchschnittlich ansteckt. Der Wert ist in Zehntelschritten von 0,0 bis 20,0 einstellbar. |
2,9 | |
| Beginn der Infektiosität: Hier lässt sich einstellen, an welchem Tag eine exponierte (nicht infektiöse) Person ins Stadium der infektiösen Erkrankung übergeht. |
6. Tag | |
| Dauer der Infektiosität: Hier kann angegeben werden, wie viele Tage die Phase der infektiösen Erkrankung andauert. |
12 Tage | |
| Dauer der Immunität: Festzulegen ist hier die Dauer der Immunität nach einer Genesung oder Impfung (in Tagen). Einstellbar sind Werte von 0 bis zu 500 Tagen oder der Wert "unendlich" (∞). |
180 Tage | |
| Sterberate: Diese Option legt fest, wie hoch der Anteil der Versterbenden unter den Erkrankten ist (0 bis 100 Prozent).  Achtung! Die hier einstellbare Sterberate ist nur ein "Richtwert". Die tatsächliche
Sterberate innerhalb eines Simulationslaufs kann zufallsbedingt leicht von der Vorgabe abweichen. Zudem erhöht sich die tatsächliche
Sterberate beim Auftreten von Infektionswellen infolge schwindender Immunität, da nun gefährdete Genesene oder Geimpfte einer
Neuinfektion und damit einem neuerlichen Sterberisiko ausgesetzt sind. Achtung! Die hier einstellbare Sterberate ist nur ein "Richtwert". Die tatsächliche
Sterberate innerhalb eines Simulationslaufs kann zufallsbedingt leicht von der Vorgabe abweichen. Zudem erhöht sich die tatsächliche
Sterberate beim Auftreten von Infektionswellen infolge schwindender Immunität, da nun gefährdete Genesene oder Geimpfte einer
Neuinfektion und damit einem neuerlichen Sterberisiko ausgesetzt sind. |
6% | |
| Therapie: Mit diesem Regler lässt sich bestimmen, in welchem Umfang der Pandemie mit medizinischen Mitteln begegnet werden kann. Zur Verfügung stehen zwei Optionen: (1) Ist der rechts neben dem Regler befindliche Schalter (siehe unten) auf "hospitalisiert" (  )
gesetzt, legt das Programm für einen Promillesatz der Bevölkerung Intensivbetten in Krankenhäusern an. Dieser Wert kann in 0,1-Promille-Schritten
von 0 bis 10 Promille gewählt werden. Angenommen wird hierbei, dass die Überlebenschancen der Patienten durch intensivmedizinische Betreuung
um einen gewissen Faktor erhöht werden können (der Faktor f = 2 bedeutet zum Beispiel, dass sich die Überlebenschance verdoppelt,
das Sterberisiko also halbiert wird; der Faktor f = 60 setzt die Überlebenschance auf das Sechzigfache). Die Höhe des Faktors hängt
vom ausgewählten Krankheitstyp ab und ist innerhalb des Programms nicht veränderbar. Bei bakteriellen Infektionen liegt der Faktor, da hier
Antibiotika verabreicht werden können, in der Regel deutlich höher als bei Viruserkrankungen, für die nicht immer ein passender Impfstoff
zur Verfügung steht. )
gesetzt, legt das Programm für einen Promillesatz der Bevölkerung Intensivbetten in Krankenhäusern an. Dieser Wert kann in 0,1-Promille-Schritten
von 0 bis 10 Promille gewählt werden. Angenommen wird hierbei, dass die Überlebenschancen der Patienten durch intensivmedizinische Betreuung
um einen gewissen Faktor erhöht werden können (der Faktor f = 2 bedeutet zum Beispiel, dass sich die Überlebenschance verdoppelt,
das Sterberisiko also halbiert wird; der Faktor f = 60 setzt die Überlebenschance auf das Sechzigfache). Die Höhe des Faktors hängt
vom ausgewählten Krankheitstyp ab und ist innerhalb des Programms nicht veränderbar. Bei bakteriellen Infektionen liegt der Faktor, da hier
Antibiotika verabreicht werden können, in der Regel deutlich höher als bei Viruserkrankungen, für die nicht immer ein passender Impfstoff
zur Verfügung steht.Infektiös Erkrankte können im Übrigen, sobald sie auf die Intensivstation aufgenommen sind, aufgrund der dort herrschenden Sicherheitsmaßnahmen niemanden mehr anstecken. Dass die Zahl der Intensivbetten nur im Promillebereich angesetzt wird, ist durchaus realistisch. Für Deutschland gilt zum Beispiel eine Intensivbetten-Bevölkerungs-Relation von ca. 0,3 Promille. Da nur rund ein Dritttel der Intensivbetten unbelegt ist, sind im Pandemiefall, bezogen auf die Gesamtbevölkerung, also nur etwa 0,1 Promille Betten verfügbar. (2) Ist der Schalter auf "ambulant" (  ) gesetzt, wird angenommen, dass die Pandemie
mit ambulanten medizinischen Mitteln (also ohne Einweisung ins Krankenhaus) zu therapieren ist. Der Regler legt nun fest, wie viele Prozent der
Gesamtbevölkerung mit einer solchen Therapie erreicht werden können. Einstellbar sind Werte von 0 bis 100 Prozent. Auch hier hängen
die Überlebenschancen der Therapierten von einem je nach Krankheitserreger unterschiedlichen Faktor f ab. Dieser ist gegenüber der Option
"hospitalisiert" allerdings nur halb so hoch, da etliche Therapiemöglichkeiten, die eine Intensivstation bietet, zu Hause fehlen. ) gesetzt, wird angenommen, dass die Pandemie
mit ambulanten medizinischen Mitteln (also ohne Einweisung ins Krankenhaus) zu therapieren ist. Der Regler legt nun fest, wie viele Prozent der
Gesamtbevölkerung mit einer solchen Therapie erreicht werden können. Einstellbar sind Werte von 0 bis 100 Prozent. Auch hier hängen
die Überlebenschancen der Therapierten von einem je nach Krankheitserreger unterschiedlichen Faktor f ab. Dieser ist gegenüber der Option
"hospitalisiert" allerdings nur halb so hoch, da etliche Therapiemöglichkeiten, die eine Intensivstation bietet, zu Hause fehlen.Zu beachten ist, dass infektiös Erkrankte in ihrem häuslichen Umfeld weiterhin andere Bewohner anstecken können, sie also durch die ambulante Versorgung nicht "neutralisiert" werden. |
0% f=2 |
|
| Therapie ambulant | hospitalisiert: Bei geöffnetem Schalter (  ) gilt die Therapieoption "ambulant", bei geschlossenem Schalter
( ) gilt die Therapieoption "ambulant", bei geschlossenem Schalter
( ) die Option "hospitalisiert" (siehe oben). ) die Option "hospitalisiert" (siehe oben). |
aus | |
| Impfquote: Über diesen Regler kann festgelegt werden, welcher Prozentsatz der Gesamtpopulation sich impfen lässt.  Diese Option ist nur aufrufbar, falls für das ausgesuchte Pathogen ein
Impfstoff zur Verfügung steht. Diese Option ist nur aufrufbar, falls für das ausgesuchte Pathogen ein
Impfstoff zur Verfügung steht. |
0% | |
| Erster Impftag: Hier lässt sich einstellen, an welchem Tag mit den Impfungen begonnen werden soll. Der Wert lässt sich in Zehnerschritten von 0 (Impfung gleich bei Simulationsstart) bis 1000 (Impfung am 1000. Tag) festlegen.  Der Einfachheit halber wird modellhaft angenommen, dass tatsächlich
alle Impfwilligen gleich am ersten Impftag geimpft werden können. Angenommen wird ferner, dass vorgenommene Impfungen
in 100 Prozent der Fälle erfolgreich sind. Sollten Sie eine Impfwirkung von weniger als 100 Prozent annehmen, müssen
Sie gegebenenfalls die Einstellung des darüberliegenden Impfquoten-Reglers nachjustieren. Der Einfachheit halber wird modellhaft angenommen, dass tatsächlich
alle Impfwilligen gleich am ersten Impftag geimpft werden können. Angenommen wird ferner, dass vorgenommene Impfungen
in 100 Prozent der Fälle erfolgreich sind. Sollten Sie eine Impfwirkung von weniger als 100 Prozent annehmen, müssen
Sie gegebenenfalls die Einstellung des darüberliegenden Impfquoten-Reglers nachjustieren. Auch diese Option ist nur aufrufbar, falls für das ausgesuchte Pathogen
ein Impfstoff zur Verfügung steht. Auch diese Option ist nur aufrufbar, falls für das ausgesuchte Pathogen
ein Impfstoff zur Verfügung steht. |
0 | |
| Mehrfachimpfungen: Der Schalter lässt in gesetztem Zustand Mehrfachimpfungen zu, falls der Immunitätsstatus der Genesenen oder Geimpften begrenzt ist.  Ist für das ausgesuchte Pathogen kein Impfstoff verfügbar, wird
der Schalter deaktiviert. Ist für das ausgesuchte Pathogen kein Impfstoff verfügbar, wird
der Schalter deaktiviert. |
aus | |
2.4 Die Zufallszahlen-NavigationDie simulierten pandemischen Verläufe basieren auf Zufallszahlen. Diese werden von PathoGen auf unterschiedliche Weise generiert:
Der Vorteil der erstgenannten Methode besteht darin, dass die so erzeugten Zufallszahlen jederzeit reproduzierbar sind. Jeder definierten Zufallszahlenkette ist eine bestimmte Laufnummer zugeordnet. So liefert beispielsweise jeder "Lauf 5" auf jedem beliebigen Rechner zu jeder beliebigen Zeit dieselben Ergebnisse, sofern die sonstigen Programmeinstellungen nicht verändert wurden; jeder "Lauf 6" führt zu neuen (anderen), aber ebenfalls spezifischen und reproduzierbaren Resultaten, jeder "Lauf 7" wieder zu neuen (anderen), aber spezifischen Resultaten und so fort. Auf diese Weise kann man bestimmte Simulationsverläufe jederzeit wiederholen und gezielt analysieren. Die zweite Methode bietet sich immer dann an, wenn man - etwa in Unterrichtssituationen oder in Seminaren - auf mehreren Rechnern zufällige Ergebnisse erzielen möchte, die sich nicht gleichen (die also wirklich "verschieden zufällig" sind). Da die Simulationsläufe niemals vollkommen identisch sind, lässt sich im Vergleich der Ergebnisse auf verschiedenen Rechnern zum Beispiel schnell klären, in welcher Bandbreite sich die Folgen bestimmter Simulationseinstellungen bewegen. In der Laufanzeige des Programms ist dieses Verfahren als "Lauf 0" bzw. als "Blackbox"-Modus gekennzeichnet (in eine Blackbox kann man nicht hineinschauen). "Blackbox"-Läufe werden nicht gezählt. Eine Durchnummerierung wäre sinnlos, da jeder Lauf einmalig und damit nicht wiederholbar ist. Die verschiedenen Zufallszahlen-Auswahlen lassen sich über vier Navigationsknöpfe steuern, die sich rechts neben den Haupttasten befinden. |
| Navigations-Knöpfe | Symbol | |
|---|---|---|
| Der erste Knopf (links) ruft den numerisch folgenden Lauf (d. h. die folgende Zufallszahlenkette) auf. Befindet sich das Programm im "Blackbox"-Modus, schaltet die Taste das PathoGen-Programm zurück in den Algorithmus definierter Zufallszahlenketten und dort in den Modus "neue definierte Zufallszahlen"; sie hat in diesem Fall die gleiche Funktion wie die Reset-Taste (siehe unten). | ||
| Der zweite Knopf (halblinks) ruft den numerisch vorangegangenen Lauf (d. h. die vorherige Zufallszahlenkette) auf. Befindet sich PathoGen im Lauf 1, schaltet die Taste das Programm in den Modus freier Zufallszahlen ("Blackbox"-Modus, "Lauf 0"). Im "Blackbox"-Modus selbst hat die Taste keine Funktion. | ||
| Der dritte Knopf (halbrechts) schaltet, sofern sich das Programm nicht im "Blackbox"-Modus befindet, zwischen
neuem Lauf (Lauf mit neuen definierten Zufallszahlen) und Laufwiederholung (Lauf mit denselben definierten Zufallszahlen) um. Der
jeweils aktuelle Modus wird in der Laufanzeige vermerkt. Der Modus "Neu" erscheint dort als dunkelroter
"New"-Kreis, der Modus "Wiederholung" als Pfeilkreis. Steht das Programm im Modus "Blackbox" (erkennbar an einer schwarzen Box in der Laufanzeige), schaltet die Taste in den Modus "Neu", also in den Modus "neue definierte Zufallszahlen", und dort zu Lauf 1.1 zurück. Dies entspricht der Funktion "Reset" (siehe unten). |
||
| Der vierte Knopf (rechts) führt einen Neustart (Reset) durch: Das Programm wird auf die Laufnummer 1.1 sowie in den Modus "Neu" zurückgesetzt; im Ergebnisfenster erscheint das Programm-Logo. Nicht zurückgesetzt werden die übrigen Programmeinstellungen, so dass mit diesen sofort weitergearbeitet werden kann. | ||
Tippt man die Vorwärts- bzw. die Rückwärtstaste schrittweise durch, wird die Laufnummer in der Laufanzeige angepasst. Gültig ist die geänderte Laufnummer allerdings erst mit dem nächsten Simulationslauf. Neue Läufe (mit neuen definierten Zufallszahlen) weist die Laufanzeige links vom Trennpunkt, Laufwiederholungen (mit denselben definierten Zahlen) in etwas kleinerer Schrift rechts vom Trennpunkt aus. |
2.5 Die Datenverarbeitungs-TastenDie mit PathoGen erzeugten Daten können im sogenannten Datenmonitor angezeigt, die hierfür relevanten Programmeinstellungen temporär oder dauerhaft gespeichert und wieder geladen werden. Das Programm stellt in diesem Zusammenhang die folgenden Tasten zur Verfügung. |
| Datenmonitor | |
|---|---|
| Die Taste öffnet ( |
|
| Über die Taste "Export" werden die Daten in ein externes (zweites) Fenster übertragen. Neben den tagesaktuellen Daten werden hier zusätzlich die Ergebnisse ausgewiesen, die auch im Ergebnisfenster zu sehen sind. | |
| Benutzerspeicher | |
| Mithilfe dieser Taste können Sie zuvor gespeicherte Benutzerdaten (siehe unten) laden. Die Daten stehen dann als "benutzerdefinierter Krankheitstyp" zur Verfügung. - Liegen noch keine Benutzerdaten vor, erscheint eine Fehlermeldung. | |
Über die fünf Schieberegler "Ausbreitungsrate", "Beginn der Infektiosität", "Dauer der Infektiosität", "Dauer der Immunität"
und "Sterberate" können Sie beliebige neue oder Varianten bestehender Pathogene definieren. Mithilfe der vorliegenden Taste lassen sich die Werte dieser
Regler sowie der Intensivmedizin-Faktor f, der Erregertyp (Virus oder Bakterium) und ein Krankheitsname speichern, und zwar
 Jede Betätigung der Taste löscht die zuvor gespeicherten benutzerdefinierten Werte des Programms
ohne Vorwarnung und ersetzt sie durch die aktuellen. Jede Betätigung der Taste löscht die zuvor gespeicherten benutzerdefinierten Werte des Programms
ohne Vorwarnung und ersetzt sie durch die aktuellen. Die Möglichkeiten zur Datenspeicherung sind vom Browsertyp und dessen Einstellungen abhängig.
Testen Sie gegebenenfalls mehrere Browser aus. Die Möglichkeiten zur Datenspeicherung sind vom Browsertyp und dessen Einstellungen abhängig.
Testen Sie gegebenenfalls mehrere Browser aus. |
|
Mit dieser Taste können Sie - nach einer Sicherheitsabfrage - dauerhaft gespeicherte benutzerdefinierte Werte
löschen, ohne sie durch neue zu ersetzen. Die temporär im Programm enthaltenen Benutzereinstellungen sind hiervon nicht
betroffen. - Liegen keine gespeicherten Benutzerdaten vor, erscheint eine Fehlermeldung. Bei Programmstart ist die Taste aus Sicherheitsgründen verborgen. Sie erscheint nur bei geöffnetem
Datenmonitor rechts unterhalb der Monitorliste. Bei Programmstart ist die Taste aus Sicherheitsgründen verborgen. Sie erscheint nur bei geöffnetem
Datenmonitor rechts unterhalb der Monitorliste. |
|
2.6 Allgemeine ProgrammfunktionenGanz oben und ganz unten auf der Benutzeroberfläche, aber auch über dem Diagrammfeld befinden sich diverse Tasten, Schalter und Regler, die nicht die Simulation selbst, sondern das Layout des Programms bestimmen bzw. externe Seiten aktivieren. |
2.6.1 Die Sprachauswahl |
| Sprachauswahl | ||
|---|---|---|
Das Programm PathoGen kann in folgenden Sprachen aufgerufen werden:
Das vorliegende Handbuch ist zurzeit nur auf Deutsch und auf Englisch verfügbar. |
||
2.6.2 Externe Seiten |
| Externe Seiten | ||
|---|---|---|
| Dieser Knopf führt zurück zur Startseite. | ||
| Über diesen Schaltknopf kann in einem separaten Fenster das vorliegende Handbuch geöffnet werden. | ||
2.6.3 Programmeinstellungen |
| Diagramm-Schalter | Startwert | |
|---|---|---|
| Mithilfe der über dem Diagrammfeld angeordneten Diagramm-Schalter lassen sich einzelne Kurven des Diagramms sowie der Impfmarker ab- bzw. zuschalten. | ||
| Gefährdete: Über diesen Schalter kann die grüne Kurve "Gefährdete" (= Gesunde, doch der Gefahr einer Ansteckung Ausgesetzte) ein- bzw. ausgeschaltet werden. |
ein | |
| Alle Fälle: Über diesen Schalter kann die schwarze Kurve "Alle Fälle" (= Summe aller bisher Exponierten bzw. Erkrankten) ein- bzw. ausgeschaltet werden. |
ein | |
| Aktuelle Fälle: Über diesen Schalter kann die rote Kurve "Aktuelle Fälle" (= aktuell Exponierte bzw. Erkrankte) ein- bzw. ausgeschaltet werden. |
ein | |
| Immunisierte: Über diesen Schalter kann die violette Kurve "Immunisierte" (= Genesene und Geimpfte innerhalb des Immunitätszeitraums) ein- bzw. ausgeschaltet werden. |
ein | |
| Verstorbene: Über diesen Schalter kann die blaue Kurve "Verstorbene" (= am Pathogen Gestorbene) ein- bzw. ausgeschaltet werden. |
ein | |
Auslastung: Über diesen Schalter kann die ockerfarbene Kurve, die den Auslastungsgrad der Krankenhaus-Intensivstationen anzeigt, ein- bzw.
ausgeschaltet werden. Die Kurve weist den Auslastungsgrad in Prozent (0 bis 100) aus. Angezeigt wird sie jedoch nur, falls der
"Therapie"-Schalter für die Simulation auf "hospitalisiert" gesetzt wurde.
Zum Vergrößern klicken Sie bitte auf die Abbildung.
Über diesen Schalter kann die ockerfarbene Kurve, die den Auslastungsgrad der Krankenhaus-Intensivstationen anzeigt, ein- bzw.
ausgeschaltet werden. Die Kurve weist den Auslastungsgrad in Prozent (0 bis 100) aus. Angezeigt wird sie jedoch nur, falls der
"Therapie"-Schalter für die Simulation auf "hospitalisiert" gesetzt wurde.
Zum Vergrößern klicken Sie bitte auf die Abbildung. |
ein | |
| Impfung: Über diesen Schalter kann der türkisfarbene Impfmarker ein- bzw. ausgeschaltet werden. |
ein | |
| Weitere Diagrammfeld-Einstellungen | Startwert | |
| Linienbreite: Hier lässt sich in einem Raster von 1 bis 10 die Breite der Linien im Diagrammfeld einstellen. - Achtung: Die Breite hat keine Auswirkungen auf den Simulationsverlauf. Sie dient lediglich der besseren Visualisierung auf kleinen Computerdisplays. |
4 | |
| Grafikgitter aus | ein: Mit dieser Option blenden Sie das Gitternetz im Diagrammfeld ein ( |
ein | |
| Auto-Reset: Ist dieser Schalter offen, bleibt jede Grafik, auch bei Ausführung des nächsten Simulationslaufs, auf dem Diagrammfeld erhalten. So kann man mehrere pandemische Verläufe übereinanderlegen und zum Beispiel die Streuung bestimmter Einstellungen ermitteln. - Bei geschlossenem Schalter wird zu Beginn jedes Simulationslaufs die vorherige Grafik automatisch gelöscht. |
ein | |
| Impfmarker: Mit diesem Schalter lassen sich Aussehen und Position der Impfmarkierung(en) auf dem Diagrammfeld bestimmen. Bei offenem Schalter werden Impfmarkierungen, ausgehend vom unteren Ende der Grafik, als senkrechter türkisfarbener Strich ( | ) gesetzt. Ist der Schalter geschlossen, erscheint stattdessen am oberen Rand der Grafik ein entsprechendes Dreieck (▼). Im Folgenden finden Sie Beispiele für beide Varianten.   Zum Vergrößern klicken Sie bitte auf die Abbildungen. |
ein | |
| Datenspeicher-Einstellungen | Startwert | |
| Datenspeicher: Ist dieser Schalter offen, lassen sich benutzerdefinierte Einstellungen nur temporär (d. h. bis zum Programmende) speichern. Bei geschlossenem Schalter können die Einstellungen auch dauerhaft auf Ihrem Rechner gesichert werden. Hierzu müssen Sie allerdings ausdrücklich Ihre Einwilligung geben. Bei erstmaligem Schließen des Schalters erscheint hierzu eine Sicherheitsabfrage. |
aus | |
| Krankheitstyp-Eingabefeld: Auf diesem Feld können Sie der benutzerdefinierten Krankheit einen Namen von maximal zwanzig Zeichen geben. Bestätigt wird die Eingabe durch einen Mausklick in das Feld oder mithilfe der Enter-Taste. Nach der Bestätigung springt das Programm auf den Krankheitstyp "benutzerdefiniert".  Das Eingabefeld ist auf zwanzig durchschnittliche proportionale Schriftzeichen ausgelegt.
Verwenden Sie viele ungewöhnlich breite Schriftzeichen, kann es passieren, dass der Name die Anzeigefenster des Programms "sprengt".
Reduzieren Sie in diesem Fall bitte die Anzahl der Zeichen. Das Eingabefeld ist auf zwanzig durchschnittliche proportionale Schriftzeichen ausgelegt.
Verwenden Sie viele ungewöhnlich breite Schriftzeichen, kann es passieren, dass der Name die Anzeigefenster des Programms "sprengt".
Reduzieren Sie in diesem Fall bitte die Anzahl der Zeichen. |
leer | |
| Datenmonitor-Einstellungen | Startwert | |
| Datenerhebung aus | ein: Ist dieser Schalter offen, erzeugt das Programm keine Daten für den Datenmonitor, das heißt, dass nachträglich keine Daten zur Detailanalyse abgerufen werden können. Sinnvoll ist das Abschalten der Datenerhebung eigentlich nur, um - zum Beispiel auf älteren Rechnern - die Rechenkapazität zu erhöhen und Berechnungen zu beschleunigen. - Bei geschlossenem Schalter ist die Datenerhebung zugeschaltet. |
ein | |
| Schriftgröße beim Datenexport: Die vom Datenmonitor generierte Tabelle kann in der Export-Fassung in verschiedenen Schriftgrößen aufgerufen werden. Die Größe lässt sich hier von 8pt bis 12pt einstellen. |
11pt | |
| Neueste Daten oben | unten: Diese Option ermöglicht das Umschalten der Richtung der Datenanzeige. Ist der Schalter offen ( |
ein | |
| Design-Einstellung | Startwert | |
| Farbwahl: Durch (wiederholtes) Drücken dieser Taste kann das farbliche Design der Benutzeroberfläche variiert werden. Es stehen zehn Farbvarianten zur Verfügung. Beachten Sie bitte, dass das tatsächlich gezeigte Design browserabhängig ist. |
1/10 | |
|
Der dem Programm zugrunde gelegte Krankheitsverlauf folgt dem SEIR- bzw. dem SEIRS-Modell zur Beschreibung der Ausbreitung ansteckender Krankheiten (SEIR: susceptible, exposed, infectious, recovered/removed, d. h. gefährdet, exponiert, infektiös erkrankt, genesen/verstorben; bei SEIRS kommt eine neuerliche suszeptible Phase hinzu). Angenommen wird also, dass sich ein Gefährdeter zunächst ansteckt, ohne infektiös zu sein (d. h. er ist lediglich "exponiert"). Nach dieser Latenzzeit gilt er, sobald er ansteckend ist, als "infektiös erkrankt" (wobei mit dieser Erkrankung Symptome einhergehen können, aber nicht müssen). Danach eröffnen sich zwei Möglichkeiten: Entweder er verstirbt - oder er gesundet nach Abklingen der Infektiosität und ist (zumindest eine Zeit lang) immunisiert (= SEIR). Schwindet seine Immunität, fällt er allerdings ins Stadium des Gefährdeten zurück (= SEIRS). Je nach Krankheitserreger variieren die Eckdaten der Erkrankung (Verbreitung, Latenzzeit, Infektiosität, Sterblichkeit, Immunität) stark. Gleiches gilt für die Erfolge intensivmedizinischer Betreuung. In der Regel ist bakteriellen Erkrankungen medizinisch (etwa durch Antibiotika) besser beizukommen als viralen Infektionen, da es gegen Letztere häufig keine adäquaten Impfstoffe gibt. Das Programm PathoGen berücksichtigt derzeit 24 verschiedene Krankheitserreger, deren Eckdaten programmseitig vorgegeben sind. Diese Daten entsprechen den aktuellen Erkenntnissen einschlägiger wissenschaftlicher Publikationen, die im Literaturverzeichnis nachgewiesen werden. Mit Blick auf künftige Virusvarianten bzw. völlig neue Virus- oder Bakterienstämme lassen sich die Werte über die entsprechenden Schieberegler jedoch weitgehend abändern bzw. anpassen. Die folgende Tabelle gibt einen Überblick über die implementierten Krankheiten und deren gesetzte Parameter. Berücksichtigt werden
|
| a | b | c | d | e | f | g | h | i |
|---|---|---|---|---|---|---|---|---|
| COVID-19 (Wildtyp) | 2.9 | 6 | 12 | 180 | 6.0 | 4 | Virus | ja |
| COVID-19 (Delta) | 6.9 | 4 | 12 | 180 | 8.0 | 4 | Virus | ja |
| COVID-19 (Omikron) | 9.5 | 6 | 12 | 180 | 1.5 | 4 | Virus | ja |
| SARS-CoV-1 2002 | 3.5 | 6 | 12 | 180 | 11.0 | 2 | Virus | nein |
| MERS-CoV 2012 | 0.8 | 10 | 10 | 200 | 34.0 | 2 | Virus | nein |
| Spanische Grippe | 2.5 | 2 | 4 | 180 | 10.0 | 2 | Virus | nein |
| Schweinegrippe | 1.5 | 2 | 4 | 180 | 0.5 | 2 | Virus | ja |
| Saisonale Grippe | 1.5 | 2 | 11 | 350 | 0.5 | 5 | Virus | ja |
| Grippaler Infekt | 2.5 | 1 | 10 | 45 | 0.0 | 1 | Virus | nein |
| Ebola-Fieber | 2.0 | 8 | 19 | ∞ | 59.0 | 10 | Virus | ja |
| Marburg-Virus | 1.3 | 6 | 16 | ∞ | 50.0 | 2 | Virus | nein |
| Lassa-Fieber | 1.5 | 8 | 15 | 30 | 5.0 | 5 | Virus | nein |
| Masern | 15.0 | 10 | 6 | ∞ | 1.0 | 4 | Virus | ja |
| Röteln (postnatal) | 6.0 | 17 | 14 | 500 | 0.0 | 1 | Virus | ja |
| Mumps | 5.5 | 17 | 14 | ∞ | 0.0 | 1 | Virus | ja |
| Windpocken | 11.0 | 15 | 10 | ∞ | 0.0 | 1.1 | Virus | ja |
| Pocken (Variola) | 6.0 | 13 | 15 | ∞ | 30.0 | 20 | Virus | ja |
| Affenpocken | 0.6 | 13 | 21 | ∞ | 1.0 | 4 | Virus | ja |
| Scharlach | 1.5 | 2 | 19 | 500 | 0.5 | 30 | Bakt. | nein |
| Keuchhusten | 6.0 | 12 | 35 | ∞ | 0.5 | 30 | Bakt. | ja |
| Diphterie 1880 | 3.0 | 4 | 21 | 500 | 50.0 | 3.3 | Bakt. | ja |
| Beulenpest | 3.0 | 4 | 4 | 180 | 60.0 | 20 | Bakt. | ja |
| Lungenpest | 3.0 | 2 | 5 | 20 | 95.0 | 4 | Bakt. | ja |
| Cholera | 9.5 | 2 | 5 | 130 | 60.0 | 60 | Bakt. | ja |
| benutzerdefiniert | 2.9 | 6 | 12 | 180 | 6.0 | 4 | Virus | ja |
|
Die Einstellung "benutzerdefiniert" (als 25. Option) entspricht bei Programmstart den Werten von COVID-19 (Wildtyp), kann jedoch beliebig geändert und gespeichert werden.
(1) Das Programm liefert keine exakten medizinischen Prognosen, sondern bildet lediglich pandemische Entwicklungstendenzen ab. Es generiert in der Regel die maximal möglichen Fallzahlen, da modellhaft angenommen wird, dass infektiöse Agenten während der gesamten Zeit ihrer Ansteckungsfähigkeit tatsächlich ungehindert ihre Mitbewohner anstecken können. In der Praxis ist dies jedoch nicht der Fall. Symptomatisch Erkrankte ziehen sich entkräftet aus der Öffentlichkeit zurück oder werden unter Quarantäne gestellt, so dass sich die Zahl ihrer Außenkontakte und damit die Weitergabe des Pathogens drastisch reduzieren. Der Pandemierechner bildet im Grunde genommen also vor allem diejenigen - tendenziell prekären - Gesellschaftsformen nach, in denen ein solcher ("freiwilliger" oder erzwungener) Rückzug nicht möglich ist. (2) Außerdem berücksichtigt das Programm als Faktor zur Dämpfung der Infektionen lediglich die ambulante oder intensivmedizinische Betreuung. Andere Faktoren zur Pandemiebekämpfung, wie etwa besondere Hygieneregeln, Reise- bzw. Kontaktverbote, die Schließung öffentlicher Einrichtungen oder gar ein kompletter Lockdown, bleiben hier ausgeblendet. Interessieren Sie sich besonders für diese letztgenannten Maßnahmen, dann konsultieren Sie hierzu bitte das themenverwandte Programm Pandemielabor. (3) Auch hinsichtlich der implementierten Impfoption steht bei PathoGen der Modellgedanke im Vordergrund. Angenommen werden nämlich zwei idealtypische Zustände: Alle gewünschten Impfungen (a) können an einem einzigen Simulationstag vorgenommen werden und erzielen (b) grundsätzlich einen hundertprozentigen Impfschutz. Auch hier sieht die Praxis leider anders aus. Immerhin kann die Effektivität der Impfungen mittelbar über den "Impfquoten"-Regler relativiert werden. (4) Das dem Programm zugrundegelegte SEIR-Modell nimmt nach einer Erkrankung eine vollständige Genesung (oder den Tod) an. Bleibende Schäden oder Folgekrankheiten, also etwa Phänomene wie Long Covid, werden hierdurch nicht abgebildet. (5) Nicht alle Infektionskrankheiten werden durch direkten Kontakt (Tröpfchen- bzw. Schmierinfektion) von Mensch zu Mensch übertragen. Viele Krankheiten, etwa Malaria, Dengue-Fieber oder Borreliose, lassen sich auf Übertragungen durch Tiere, etwa Mücken und Zecken, zurückführen. Die letztgenannten Krankheiten sind nicht in das vorliegende Programm aufgenommen, da hierzu völlig andere Algorithmen (die z. B. die für den Lebensraum der Tiere unabdingbaren klimatischen und saisonalen Verhältnisse berücksichtigen) benutzt werden müssten. (6) Eine Ausnahme bildet hier lediglich die (im Programm enthaltene) Beulenpest, da deren Überträger (Rattenflöhe) unabhängig von Klima und Region in enger Symbiose mit dem Menschen leben und dadurch eine unmittelbare Ansteckung beispielsweise über menschliche Kleidung (also Quasi-Körperkontakt) möglich ist. (7) Als problematisch erweist sich die Simulation der (hier ebenfalls aufgenommenen) Cholera. Eine Modellierung dieser Krankheit mithilfe des SEIR(S)-Modells ist nur bedingt möglich, da die Übertragung eher selten direkt von Mensch zu Mensch, in der Regel hingegen indirekt über verunreinigtes Wasser, Exkremente oder kontaminierte Lebensmittel erfolgt. Eine SEIR(S)-kompatible Modellierung, so wie sie hier vom Programm vorgenommen wird, gilt eigentlich nur für Populationen in dezidiert prekären Lebensverhältnissen (Zusammenleben auf sehr engem Raum unter schlechten hygienischen Bedingungen). (8) Nicht aufgenommen sind die wichtigen Krankheiten Tuberkulose und HIV/AIDS, da deren Infektionsverlauf sich einer einfachen Modellierung im SEIR(S)-Modell entzieht. Im Fall der Tuberkulose kann die Latenzzeit einige Wochen oder Monate betragen, sich aber auch über mehrere Jahre erstrecken, bei den meisten Exponierten (etwa 9 von 10) bricht die Krankheit sogar nie aus. Ähnliches gilt für HIV; hier kommt noch hinzu, dass die Patienten in der Regel nicht an HIV selbst, sondern an einer von zahlreichen (diffusen, hier nicht modellierten) Folgeerkrankungen sterben (z. B. an mykobakteriellen Infektionen [B20.0] wie Tuberkulose oder an Pneumocystis-Pneumonie [B20.6]). (9) Nicht für alle der in der obigen Tabelle aufgeführten Krankheiten stehen verlässliche, eindeutige Daten zur Verfügung. Diese variieren zum Teil je nach Untersuchungszeitraum und Region beträchtlich. Für historische Pandemien (Beulenpest, Diphterie der 1880er Jahre, Spanische Grippe usw.) liegen ohnehin nur Schätzwerte vor. So schwanken beispielsweise die Schätzungen zur Zahl der durch die Spanische Grippe (1918-1920) bedingten Todesfälle, je nach wissenschaftlicher Publikation, zwischen 20 und 50 Millionen weltweit. Die im Programm benutzten Zahlen stellen daher teils nur Näherungswerte dar. (10) Zum Teil variieren die für eine Krankheit ermittelten Eckdaten auch deshalb stark, weil selbstverständlich nicht alle Patienten in gleicher Weise auf einen Erreger reagieren. Daher sind in der Tabelle bzw. im Programm häufig (gewichtete) Mittelwerte gesetzt. So liegt die Latenzzeit zum Beispiel bei COVID-19 (Wildtyp) in einem Spektrum von zwei bis vierzehn Tagen, typischerweise setzt die infektiöse Erkrankung allerdings nach knapp einer Woche ein. Der Beginn der Infektiosität ist deshalb in der Tabelle mit dem "starren" Wert 6 (= 6. Tag) angegeben und wird im Programm auch ebenso verrechnet. (11) Der Einfachheit halber setzt das in PathoGen verwendete Modell die Immunität Genesener und Geimpfter gleich. In der Praxis kann es hier jedoch deutliche Abweichungen geben. So sind Patienten nach einer tatsächlichen Infektion mit Masern oder Mumps in der Regel ein Leben lang immun; einen (annähernd) vergleichbaren Schutz erzielt man jedoch erst mit einer Zweifachimpfung. (12) Für manche, vor allem neuartige Erkrankungen wie COVID-19 lässt sich die Dauer der anschließenden Immunität (noch) nicht genau festlegen. Gelegentlich wird eine bestehende Immunität durch mutierte Varianten des Erregers ausgehebelt. Unter Berücksichtigung dieser Varianten nimmt das Programm zum Beispiel für SARS-CoV-2 derzeit eine Immunität von etwa einem halben Jahr (180 Tage) an. Auch der diffuse grippale Infekt (mit über 200 Virusvarianten) erfordert eine jährlich neu ausgerichtete Impfung. (13) Die Effektivität einer intensivmedizinischen Betreuung ist in einigen Fällen schwer zu ermitteln, da für medizinisch gut versorgte Regionen nur gemilderte, nicht aber "natürliche" Sterbeziffern vorliegen. Um die Effektivität von Intensivbehandlungen zu messen, müsste man einer Gruppe von Versorgten eigentlich eine Kontrollgruppe nicht betreuter Patienten gegenüberstellen. Dies ist natürlich aus ethischen Gründen nicht zu verantworten. (14) Mitunter konfligieren auch medizinische und juristische Angaben. So wurde im frühen 20. Jahrhundert beispielsweise gegen die Beulenpest geimpft; auch heute noch werden (in ihrer Wirkung umstrittene) Pest-Schutzimpfungen angeboten. Das Robert Koch-Institut meldet aktuell hingegen, dass es derzeit keinen zugelassenen Pest-Impfstoff gebe. (15) Die Software PathoGen erhebt keinen Absolutheitsanspruch. Falls Ihnen andere medizinische Eckdaten als die im Programm vorgegebenen vorliegen, sollten Sie nach Belieben von der Möglichkeit Gebrauch machen, die Werte über die Stellschrauben (Regler, Schalter) anzupassen. |
Im folgenden Abschnitt finden Sie einige technische Hinweise, die für die optimale Nutzung des Programms hilfreich sein können. 4.1 SystemvoraussetzungenPathoGen ist in JavaScript programmiert und daher auf vielen Plattformen lauffähig. Erfolgreich getestet wurde das Programm unter MS Windows (TM), macOS (TM) und Android (TM) auf folgenden Browsern:
Bekannte Einschränkungen:
4.2 Bekannte Probleme(1) Auf älteren Rechnern sollten Sie die Population nicht gleich auf die höchste Stufe (1 Million) setzen, da die Hardware die komplexen Berechnungen des Programms sonst unter Umständen nur mit beträchtlichem Zeitaufwand oder aber gar nicht mehr bewältigt. Starten Sie also mit einer moderaten Personenzahl (Programmvorgabe: 1000) und testen Sie die Möglichkeiten Ihres Computers schrittweise aus. (2) Eine problemlose, über die reine Anzeige hinausgehende "echte" Übertragung der Daten per Datenexport ist derzeit nur mit dem Firefox möglich. In den übrigen Browsern wird die formatierte Export-Tabelle zwar ordnungsgemäß in einem externen Fenster geöffnet, jedoch lässt sie sich hieraus nicht oder nur auf Umwegen in ihrer ursprünglichen Formatierung per Copy & Paste in externe Programme wie MS Word (TM) übernehmen. (3) Sollte die Simulation auf einem älteren Rechner oder in bestimmten Netzwerkumgebungen nicht zufriedenstellend laufen, können Sie, falls Sie sie nicht benötigen, die Datenermittlungs-Option abschalten. Dadurch wird programmintern die Routine zur Bereitstellung der Monitordaten deaktiviert, was wiederum Rechenkapazität einspart und so den Lauf vielleicht verbessert. (4) Die Möglichkeit, Simulationsdaten dauerhaft auf dem eigenen Rechner zu speichern, hängt vom verwendeten Browsertyp und dessen Einstellungen ab. Gespeichert wird nicht über Cookies, sondern über die Javascript-Funktion localStorage. Im Lauf der Zeit haben einige Browser das Handling dieser datenschutzsensiblen Funktion bereits geändert; weitere Änderungen sind nicht auszuschließen. 4.3 Häufig gestellte FragenAuch wenn ich die Einstellungen des Programms beibehalte, zeigen die einzelnen Simulationsläufe unterschiedliche Ergebnisse. Wie ist das zu erklären? Die Ergebnisse belegen, dass die Wirklichkeit nicht komplett berechenbar ist. Das Programm liefert lediglich Wahrscheinlichkeiten, dass sich die Resultate innerhalb eines bestimmten Spektrums bewegen. Mich irritiert, dass die Simulationsergebnisse wiederholbar sind. Handelt es sich also gar nicht um "zufällige", sondern um "programmierte", womöglich manipulierte Resultate? Alle Simulationsergebnisse folgen dem Zufall. Den Modus, in dem definierte Zufallszahlenketten verwendet werden, sollte man sich nicht so vorstellen, dass dort fertig konzipierte Zahlen vorliegen; vielmehr werden diese sehr komplex erzeugt und divergieren bereits bei minimal veränderten Simulationseinstellungen. Der Terminus "Zufallszahlenketten" beschreibt also eher ein vieldimensionales Geflecht von Zufallszahlen. Manipuliert wird hier nichts. Die Berechnung der Zufallszahlen folgt einem Algorithmus von Makoto Matsumoto, Takuji Nishimura und Sean McCullough. Der Quellcode zum Programm PathoGen liegt offen; die Programmierung ist also im Detail überprüfbar. - Die Ergebnisse, die im Modus "zufällige Zufallszahlen" erzeugt werden, weichen im Übrigen nicht von denen im Modus der definierten Zufallszahlen ab. Ist das dem Programm zugrunde liegende Modell nicht allzu simpel? In der Realität hängt die Verbreitung von Infektionen doch von vielen verschiedenen Faktoren ab, etwa dem Alter der beteiligten Personen, ihren Bezugsgruppen, der Bevölkerungsdichte allgemein, vor allem aber auch der medizinischen Infrastruktur. Das ist korrekt. PathoGen basiert auf einem sehr einfachen Modell mit sehr reduzierten Infektionswegen. Eine komplexere Simulation, die Ihre Einwände berücksichtigt, finden Sie zum Beispiel in dem Pandemie-Programm Pandemielabor. Der benutzerdefinierte Krankheitstyp ist standardmäßig auf das Merkmal "Virus" eingestellt. Wie kann ich dies in "Bakterium" ändern? Ganz einfach. Wählen Sie ein bakterielles Pathogen, also zum Beispiel Diphtherie, Beulenpest oder Cholera, passen Sie dort die Einstellungen Ihren Vorstellungen an und speichern Sie dann das Ganze (via "Speichern"-Taste). Anschließend können Sie Ihr neu erstelltes Pathogen als Bakterium über die Option "benutzerdefiniert" aufrufen. Wie lässt sich der Intensivmedizin-Faktor an meine Vorstellungen anpassen? Nur begrenzt. Wählen Sie ein voreingestelltes Pathogen, dessen Faktor Ihren Vorstellungen am ehesten entspricht, und passen Sie dann die Einstellungen dieses Krankheitstyps an Ihre Erfordernisse an. Den Faktor selbst können Sie leider nicht verändern. Ein Faktor f = 60 lässt sich also über das Pathogen Cholera sehr wohl erzeugen, ein Faktor f = 23 oder f = 9 allerdings nicht. Wenn ich die intensivmedizinische Therapie zuschalte, kann es passieren, das während des gesamten Simulationslaufs gar niemand erkrankt. Wie ist das möglich? Liegt die Zahl der Exponierten bei Programmstart niedriger als die Zahl der zur Verfügung gestellten Intensivbetten, werden alle Infizierten natürlich sofort bei Ausbruch der Infektiosität ins Krankenhaus aufgenommen. Dort können sie dann niemanden mehr anstecken. Die Pandemie ist damit beendet, bevor sie überhaupt begonnen hat. Jedesmal, wenn ich einer benutzerdefinierten Krankheit, die auf einem vorprogrammierten Pathogen basiert, einen Namen geben will, springt das Programm auf den Krankheitstyp "benutzerdefiniert" und meine Reglereinstellungen gehen verloren. Was ist zu tun? Speichern Sie zunächst Ihre individuellen Reglereinstellungen, geben Sie dann erst den Namen der Krankheit ein und speichern Sie schließlich das Ganze erneut. Nun dürfte alles gesichert sein. Meine Benutzerdaten lassen sich nicht dauerhaft speichern, obwohl ich den "Datenspeicher"-Schalter gesetzt habe. Woran kann das liegen? Das kann mehrere Gründe haben. Gespeichert werden die Daten zwar nicht über Cookies, sondern über die Javascript-Funktion localStorage. Vielleicht ist Ihr Browser jedoch so eingestellt, dass er sowohl die Nutzung von Cookies als auch die von localStorage verbietet. In diesem Fall müssen Sie die Einstellungen Ihres Browsers ändern (z. B. Cookies erlauben). Unter Umständen blockiert Ihr Browser auch das Handling von localStorage in bestimmten Arbeitsumgebungen, zum Beispiel, wenn Sie das Programm PathoGen lokal auf Ihren Rechner heruntergeladen haben und offline benutzen. Probieren Sie bei Schwierigkeiten bitte verschiedene Browsertypen aus. Welche zusätzlichen Funktionen sind in Zukunft für das Programm geplant? PathoGen ist bewusst einfach gehalten und modelliert pandemische Geschehnisse nur sehr allgemein. Dabei soll es auch bleiben. Falls Sie sich für komplexere Pandemie-Simulationen interessieren, nutzen Sie bitte das Pandemielabor. Dieses bietet sehr ausgefeilte Einstellmöglichkeiten und noch erheblich umfangreichere Optionen zur Datenanalyse. |
Eine Benachrichtigung des ursprünglichen Autors bei nicht-trivialer Modifizierung des Programms ist ausdrücklich erwünscht. Änderungen am Text des Handbuchs bitte nur nach Rücksprache mit dem Autor. Bei Rückfragen wenden Sie sich bitte an folgende Adresse:
|
| Deutscher Text: Frank U. Kugelmeier, Attendorn Version 3 |

 |
Zum Anfang der deutschen Dokumentation. |
 |
To the beginning of the English documentation. |
 |
Cette documentation n'est actuellement disponible qu'en allemand et en anglais. |
 |
Esta documentación sólo está disponible actualmente en alemán e inglés. |
 |
Esta documentação está actualmente disponível apenas em alemão e inglês. |
 |
Questa documentazione è attualmente disponibile solo in tedesco e in inglese. |
 |
Deze documentatie is momenteel alleen beschikbaar in het Duits en Engels. |
 |
Dokumentacja ta jest obecnie dostępna tylko w języku niemieckim i angielskim. |
 |
Bu belge şu anda yalnızca Almanca ve İngilizce olarak mevcuttur. |
 |
Ĉi tiu dokumentaro nuntempe disponeblas nur en la germana kaj la angla. |
